1Халдварт Өвчин Судлалын Үндэсний Төв
2Монгол Улсын Их Сургууль
3ОХУ-ын ШУА-ийн Молекул-генетикийн хүрээлэн
4АНУ-ын Өвчний хяналт сэрийлэлтийн төв
This study analyzed 140 varicella zoster virus (VZV) samples collected in 2004- 2009 from patients hospitalized in the National Center of Communicable Diseases, Ulan-Bator, Mongolia with clinical diagnosis of varicella. The method based on amplification of specific DNA fragments of the ORF22 gene was used, followed by the sequencing and detection of the status of characteristic point mutations at positions 37902, 38055, 38081 and 38177 in this fragment. The results indicated that the collected samples belonged to genotypes J (59,3%), M1 (22,0%), E1 (10,2%), E2 (5.1%), and M2 (3.4%). ORF 62 for the cleavage site SmalI and ORF 38 and ORF 54 for the cleavage sites Pstl and Bgll were also analyzed. All the samples were Sma-. All samples with genotype E were PstI+ BglI-; all samples with genotype M1 and M2 were PstI+ BglI+. Out of 35 samples with genotype J, 33 and 2 were PstI+ BglI+ and PstI- BglI+, respectively. The study could identify the genotypes of VZV circulating in Mongolia and confirmed the absence of mutations characteristic for the vaccine strain. Our results have shown VZV strains circulating in Mongolia is extremely heterogeneous and may shed new lights on the current understanding of VZV evolution.
Mongolian Journal of Infectious Diseases Research, 2011, No1 (38):3-7; 1table, 8 figures, 16 references
Салхинцэцэг бол амьсгалын замаар халдан тархдаг, хүүхдийн насны цочмог халдварт өвчин бөгөөд улаанбурхан зэрэг өвчнийг дархлаажуулалтаар хяналтанд авахын хэрээр Монгол улсад бүртгэгдэж буй халдварт өвчний дотор хувийн жин нь ихэсч, ойрын ирээдүйд ихсэх хандлагатай халдварт өвчний тоонд орох болж байна [1]. Энэ өвчний үүсгэгч нь херпесвирүсийн овогт багтдаг бөгөөд анхны халдварын дараа мэдрэлийн ширхэгт бүгж хоцроод, хожим дархлаа сулрахад илрэн гардаг [2] хэмээн нийтээр хүлээн зөвшөөрөх болоод байгаа бүслэх үлд (зостер, буюу “могой яр”) хэмээх эмнэлзүйн хэлбэрийг нь харгалзан салхинцэцэг-бүслэх үлдийн вирүс (СБВ) гэж нэрлэдэг. СБВ-ийн удамшлын хэвшинжийг мэдрэг, өвөрмөц чанар өндөртэй молекул биологийн аргаар судлах нь халдварын хэлхээг нарийвчлан тогтоож, зэрлэг омгийн вирүсийг вакцины омгоос ялгах практикийн ихээхэн үр өгөөжтэй. Харамсалтай нь манай улсад энэ талын судалгаа хомс байгаа бөгөөд бид 2004 оноос энэ талын судалгааг эхэлж, урьдчилсан дүнг нийтлүүлж байсан билээ [3, 4].
Одоогийн байдлаар СБВ-ийн 22 омгийн нүклеотидийн бүрэн дарааллыг тогтоогоод байна [5]. Эдгээрийн болон бусад зарим омгийн хэсэгчилсэн нүклеотидийн дарааллын судалгаа нь СБВ–ийг 5 генотип (удамшлийн хэвшинж): Европ 1 (E1), Европ 2 (E2) , Япон (J), Холимог 1 (M1), Холимог 2 (M2) болгон хуваах бололцоо олгож байгаа юм [6].
Бид Монгол улсад орчиж буй СБВ-ийн хэвшинжүүдийг ORF22-ын нүклеотидийн дарааллын өвөрмөц байдлаар болон вакцины омгийн вирүс эргэлтэнд байгаа эсэхийг нь рестрикцийн фрагментын уртын полиморфизмийн (РФУП) аргаар тогтоох зорилго тавьж энэ судалгааг гүйцэтгэв.
Судалгааны хэрэглэгдэхүүн
Уг судалгаанд 2008-2009 онуудад салхинцэцэг гэсэн эмнэлзүйн оноштойгоор халдвар авч ХӨСҮТ-д хэвтэн эмчлүүлж байсан өвчтнүүдээс манай улсад бүртгэгдэж буй өвчлөлийн бүтцийг тусгасан санамсаргүй түүврийн аргаар 140 өвчтөн сонгов [Хүснэгт 1].
Хүснэгт 1. Судалгаанд авсан өвчтөний нас, хүйсийн бүтэц
Эдгээр өвчтөний цэврүүний шингэнийг нь 0.05 М PBS-ийн уусмалд хийх буюу Whatman FTA-ийн цаасан дээр шингээн авч хадгалсан. ДНХ ялгах Whatman FTA-ийн цаасан дээр шингээн авч хадгалсан вирүсийн ДНХ-г үйдвэрлэгчийн зааварын дагуу ялгав. 0.05 М PBS-ийн уусмалд хийсэн цэврүүний шингэнээс вирүсийн ДНХ-г Qiagen QIAamp DNA Mini Kit ашиглан үйлдвэрлэгчийн зааврын дагуу ялгав.
УТ-ПГУ
Сорьцийг хүний эд эснээс зөв авсаныг хүний генийн экпрессийн хяналт болгон ашигладаг GAPDH генийн mRNA-ийн экспрессээр тодорхойлов. УТ-ПГУ-г бодит хугацааны ПГУ-ийн машин (Stratagene Mx 3005P, АНУ)-д олшруулж шинжилгээний дүнг гаргав. Урвалын холимог болон олшруулалтыг Барбер нарын аргаар [7] гүйцэтгэв.
ПГУ
СБВ-ийг илрүүлэх ПГУ-ыг MWG Bioneer, Primus 96-plus (Korea) ПГУ-ийн машин ашиглан Лопарёв нарын боловсруулсан аргаар [8] гүйцэтгэв.
ORF62-ын SmaI рестрикцийн сайт илрүүлэх шинжилгээ
Вирүсийн зэрлэг омийг вакцины омгоос ялгах шинжилгээг MWG Bioneer, Primus 96-plus (Korea) ПГУийн машин ашиглан Лопарёв нарын боловсруулсан РФУП-ийн аргаар [9] хийв.
ORF38-ын PstII, ORF54-ийн BglI сайт илрүүлэх шинжилгээ
PstII, BglI сайт дахь удамшлын хэвшинжийг тодорхойлох шинжилгээг MWG Bioneer, Primus 96-plus (Korea) ПГУ-ийн машин ашиглан ЛаРусса нарын боловсруулсан РФУП-ийн [10] аргаар хийв.
Нүклеотидын дарааллыг тогтоох шинжилгээ
СБВ-ийн ORF22 генийн нүклеотидын дарааллыг тогтоохын тулд Qiagen PCR цомгоор ORF22 R1f, ORF22 R1r праймерууд [8] ашиглан MWG Bioneer, Primus 96-plus, Korea 446 хос нуклеотидийн урттай бүтээгдэхүүн гаргаж авав.
ПГУ-ын бүтээгдэхүүнийг Bioneer, Korea, Accu-Prep PCR purification цомог ашиглан үйлдвэрлэгчийн зааврын дагуу цэвэрлэв. Цэвэрлэсэн ПГУ-ын бүтээгдэхүүнд Applied Biosystem BigDye Terminator V3.1 Cycle Sequiencing цомог ашиглан ПГУ [5] тавив. Нуклеотидийн дараалалыг ABI 3130 xl Genetic Analyzer-аар тодорхойлов.
Био-информатикийн анализ-ыг ABI-ийн SeqScape v2.5 программ, MEGA 4 программыг тус тус ашиглан гүйцэтгэв.
Үр дүн
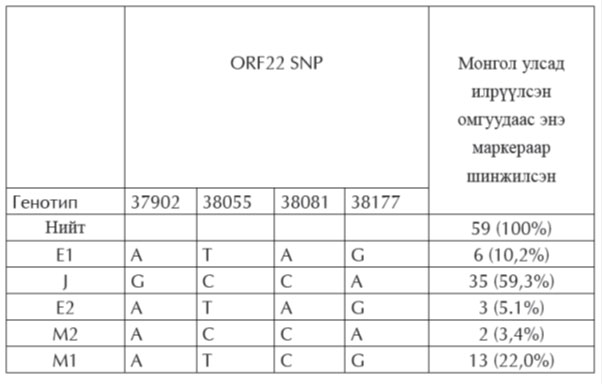
Бодит хугацааны УТ-ПГУ-аар GAPDH-ийн mRNA 139 (99,2%) сорьцонд илэрсэн [Зураг 1]. Энэ нь сорьц зөв авагдсаныг гэрчилж буй юм.
Зураг1. GAPDH генийн праймер ашиглан явуулсан бодит хугацааны УТ-ПГУ-ын зураг
Электрофорезоор ORF22-ын ПГУ-ын бүтээгдэхүүний эерэг ба сөрөг хяналт зөв явагдсан бөгөөд цэврүүний шингэнээс ялгасан ДНХ нь сонгон авсан хяналтын ДНХ-тай адил зураглал үүсгэсэн байв [Зураг2].
ПГУ-аар ORF22-ын шууд ба урвуу праймер ашиглан СБВийн ДНХ илрүүлэхэд нийт 139 сорьцноос 131 (94,2%) тохиолдолд илэрсэн байв. 8 сорьцонд (803047, 0405023, 0412002, 2, 0508046, 0508047, 0509049, 0510051 дугаартай сорьц) СБВ-ийн ДНХ илрээгүй байна. Зарим сорьцонд СБВ илрээгүй нь сорьцыг хожуу хугацаанд авсантай холбоотой байж болох юм.
Зураг 2. 0805073-0808101 дугаартай соьцноос ялгасан ДНХ-г ORF22 генийн хэсгийг ПГУ-аар олшруулсан зураг. М-size marker.
Вирүсийн зэрлэг омгийг вакцины омгоос ялгах шинжилгээг РФУП-ын аргаар 50 сорьцонд хийв. Вакцины омгийн хяналтын бүтээгдэхүүнийг SmaI рестриктазаар зүсэхэд 112, 79, 41 ба 36 хос нуклеотидын урттай дөрвөн хэрчим үүсэж байсан бол өвчтний сорьцноос ялгасан ДНХ-д 153, 79 ба 36 хос нуклеотидын урттай гурван хэрчим үүссэн байна [Зураг 3]. Санамсаргүй аргаар сонгосон 50 сорьцонд вакцины омгийн ДНХ өвөрмөц SmaI рестрикцийн сайт илрээгүйгээс үндэслэн Монгол улсад орчиж буй СБВ нь зэрлэг омгийх болох нь тогтоогдов. Иймд бусад сорьцонд дээрх шинжилгээг хийх шаардлагагүй гэж үзэв
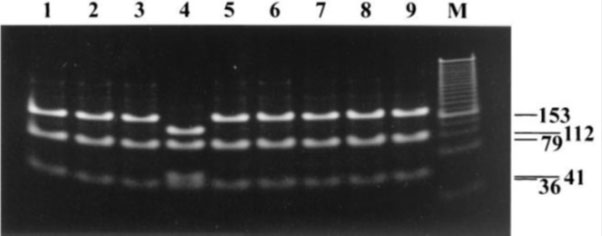
Зураг 3. PKVL6U ба PKVL1L праймераар олшруулсан ORF62 генийн хэсгийг SmaI рестриктазаар зүссэн зураг, М-size marker, 1-3 -0412001-0412003 дугаартай сорьц, 5-9- 0412005-0412009 дугаартай сорьц.
ORF38-ын PstII, ORF54-ийн BglI сайт илрүүлэх шинжилгээг мөн РФУП-ын аргаар хийж генотипийг тодорхойлов. 804050, 805061, 901009 дугаарын сорьцноос ялгасан ДНХ-ийн ORF38 гений хэсгийг ORF38 PstI A ба ORF38 PstI В праймераар, ORF54 гений хэсгийг ORF54 Nla ба ORF54 Fok праймераар олшруулах шинжилгээ огт явагдаагүйгээс дахин тавив.
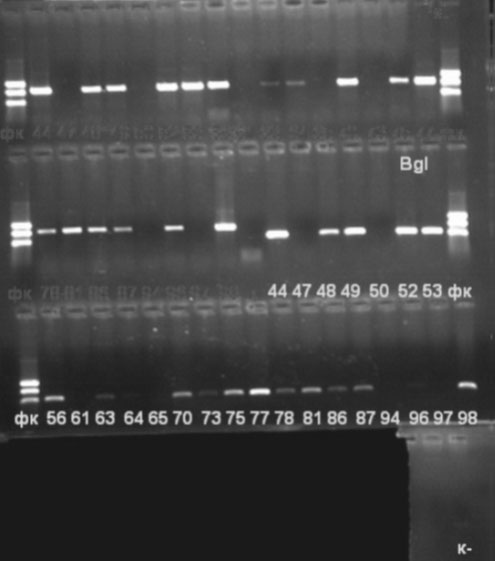
Зураг 4. Зарим сорьцны ДНХ-г ORF38-ийн Lar-3 ба Lar-4, ORF54-ийн Lar-1 ба Lar-2 праймераар олшруулж авсан бүтээгдэхүүний зураг, Фк-size marker.
Давтан шинжилгээгээр ПГУ явагдаагүй учир [Зураг 4] эдгээр сорьцийг РФУП-ын шинжилгээнд оруулах боломжгүй болов. Эдгээр сорьцны олшруулалт явагдаагүй шалтгаан нь ялгасан ДНХ хадгалах явцад задарсантай холбоотой хэмээн үзэж байна. 805063, 805064, 805065, 805073, 806078, 807094 дугаарын 6 сорьцны ORF38 PstI A ба ORF38 PstI В праймераар ДНХ-ийн хэсэг олшрогдсон боловч ORF54 Nla ба ORF54 Fok праймераар ДНХ-ийн хэсэг олшрогдоогүй байна. Өөрөөр хэлбэл эдгээр сорьц бүгд PstI+ боловч BglI сайт нь тодорхойгүй байна. PstI+ байгаа учир эдгээр сорьц бүгд зэрлэг омгийх гэж урьдчилан хэлж болох боловч олон улсын практикт зэрлэг, вакцины омгийг ялгах болон хэвшинжийг тодорхойлоход PstI болон BglI рестрикцийн сайт хосоороо байж маркер болдог. Иймд энэхүү зургаан сорьцийн хэвшинжийг BglI рестрикцийн сайтын мэдээлэл үгүй учраас хэлэх боломжгүй болсон. Эдгээр сорьцонд бидний сонгосон ORF54 Nla ба ORF54 Fok праймер ажиллахгүй байгаа нь урьд өмнө нь тэмдэглэгдээгүй SNP тухайн хэсэгт байж болзошгүй хэмээн үзэж цаашид судлах шаардлагатай байна.
Нийт 131 сорьцноос 9 (6,9%) сорьцонд ПГУ-ын олшруулалт болон ба BglI-ийн рестрикци нь яваагдаагүйгүйгээс эдгээр 9 сорьцийг энэхүү шинжилгээнээс хассан боловч ийм төрлийн тохиолдлыг цаашид нарийвчлан судлах шаардлагатай юм.
Үлдсэн 122 сорьцноос 12 (9,8%) сорьц PstI+Bgl-, 3 (2,4%) сорьц PstI –Bgl+, 107 (87,7%) сорьц PstI+Bgl+ байв [Зураг 5,6].
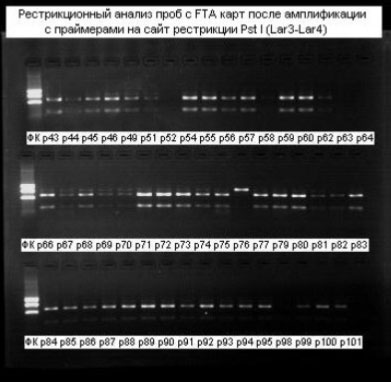
Зураг 5. PstII рестрикцийн бүтээгдэхүүний зураг (0803043-0808101 дугаар сорьц), Фк-size marker.
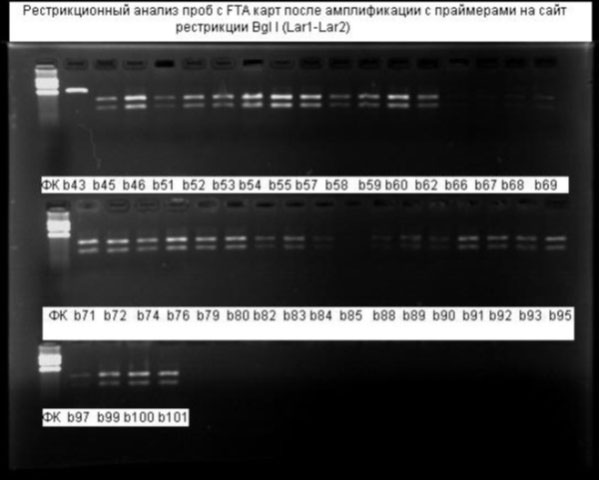
Зураг 6. BglI рестрикцийн бүтээгдэхүүний зураг (0803043-0808101 дугаар сорьц), Фк-size marker.
СБВ-ийн нүклеотидын дарааллыг тогтоох шинжилгээг өөрийн орны нөхцөлд бие даан хийв [Зураг 7].
Зураг 7. Нүклеотидын дарааллыг тогтоох шинжилгээний явц
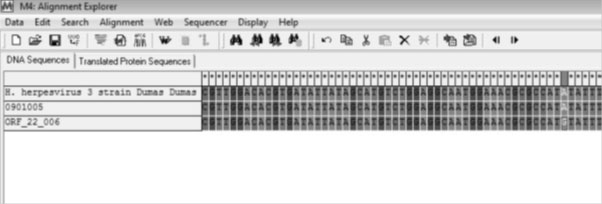
Зураг 8. Монголд илрүүлсэн СБВ-ийн ORF22-ийн нуклеотидудын дарааллыг Dumas омгийн ORF22-ийн нуклеотидтай Mega 4 програмаар харьцуулсан дүн. Вирүсийн генотипийг ORF22-ын 37902, 38055, 38081, 38177 дахь SNP-ээр тодорхойлов (Зураг 8), [8].
Хүснэгт 2. ORF22-ын 4 SNP-ээр тодорхойлсон генотипийн Тархалт
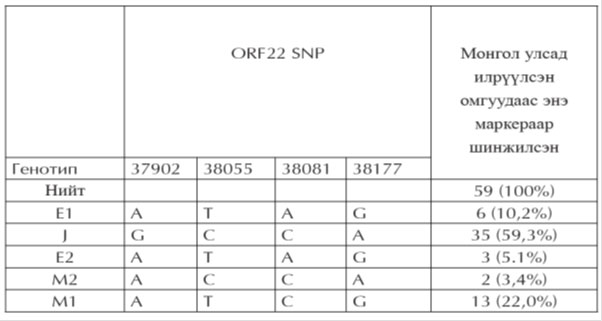
Ийнхүү Монголд илрүүлсэн 59 СБВ-ийн 35 (59,3%) нь J, 13 (22,0%) нь M1, 3 (5.1%) нь Е2, 6 (10,2%) нь Е1, 2 (3,4%) нь M2 байв. Эдгээрээс 9 вирүсийн ORF22 хэсэгчилсэн дарааллыг GenBank-нд HQ630845-HQ63053 дугаараар бүртгүүлэв.
Хэлцэмж дүгнэлт
Монгол улсад орчиж буй СБВ-ийн генотипийг Лопарёв нарын [6] боловсруулсан аргаар тодорхойлоход 7 генотипийн 5 буюу J, M1, М2, Е1, Е2 генотипийн вирүс Монгол улсад идэвхитэй орчиж байна. Нийт илэрсэн вирүсүүдээс ихэнх (59,3%) нь J генотипийх байв. J генотип голдуу Япон, Австрали, Зүүн өмнөд Азид тархсан байдаг [6]. Европ болон Хойд Америкд илрүүлсэн СБВ-т J генотип огт илрээгүй байна [11,12]. Өмнөд Солонгос, Хятадад J генотипийн вирүс өргөн тархалттай байв [13, 14]. J генотип Японд давамгайлан тархсан учир Японоос гаралтай гэж үзэж [6] байсан. Харин манай судалгаа энэхүү генотип Төв болон Зүүн өмнөд Азид үүсэн, бусад нутаг дэвсгэрт тархсан байж болзошгүй гэсэн шинэ таамаглалын үндэслэл болж байгаа юм. Манай судалгаагаар J генотипээс гадна М1 (22%) генотип илэрсэн байна. М1 генотипийн омгууд Африк, Хойд Америк, Өмнөд Америк, Энэтхэг, Хятадын Өмнөд хэсэг, Зүүн Өмнөд Азид давамгайлан тархсан байдаг [5]. Монголд илрүүлсэн хоёр вирүсийн хэвшинж нь M2 байв. Гэхдээ энэ 2 вирүс Америкт голчлон орчиж буй омгуудынхаас [4] зарим цэгчилсэн мутацаар өөр байсан нь цаашид гүнзгийрүүлэн судлах шаардлагатай болгож байна. Мөн Европд л тохиолддог, гэхдээ ховор илэрдэг Е1, Е2 генотип [15] бидний судалгаагаар Монголд илэрсэн нь ихээхэн сонирхолтой байгаа болно. Монголд СБВийн Азийн орнуудад давамгайлан тархсан J голлон тархсан байгаа боловч, дэлхийн өөр бүх нутгуудад л олддог М1, М2, Е1, Е2 генотип илэрсэн, мөн одоогоор ангилалд хамааруулж болохгүй байгаа омгууд илэрсэн нь Монгол улсад СБВ-ийн олон генотип зэрэгцэн орчилд байгааг нотлож байгаа бөгөөд СБВ-ийн эволюци, тархалтын түүхэн замналын тухай одоогийн тогтсон төсөөллийг өөрчлөхөд хүргэж болзошгүй гэж үзэхэд хүргэж байна.
Орчилд байгаа зэрлэг омгийн вирүсийг вакцины омгоос ялган салгахдаа ORF38 болон ORF54 –ийн PstII ба BglI рестрикцийн сайт, ORF62-ын SmaI рестрикцийн сайтын РФУП-ын аргыг хэрэглэсэн билээ. Мөн бидний судласан 6 (4,7%) омог одоо генетипийн маркер болгон ашиглаж буй рестриктазаар зүсэгдэхгүй байгаа нь судлаачдын урьд бичиглээгүй генотип ч байж болзошгүй болгож байна. РФУП-ын аргаар шинжилсэн нийт 122 сорьц зэрлэг омог бөгөөд тэдгээрийн 87,7% нь PstII+/BglI+, 9,8% нь PstII+/BglI- буюу зэрлэг омгийн хэвшинжтэй байв. Харин 2,4% нь PstII- BglI+ буюу вакцины хэвшинжтэй ижил байв. Ийм хэвшинжтэй зэрлэг вирүс эргэлтэнд байгааг эрдэмтэд тэмдэглэсэн байдаг [6]. Гэхдээ эдгээр омгийг ORF62-ын SmaI сайтаар судлахад Sma- буюу зэрлэг омгийх болохыг тогтоов. PstII ба BglI рестрикцийн сайт нь СБВ-ийн тодорхой улс орон болон нутаг дэвсгэрт дагнан буюу давамгайлан тархсан байдлыг судлах хэвшинжийн маркер болдог. Манай судалгаагаар илрүүлсэн СБВ-ийн дийлэнх (107 буюу 87,7%) нь PstII+ BglI+ байсан нь энэ хэвшинж Азид илүү тархалттай байдаг тухай бусад судлаачдын [16] дүгнэлтийг баталж байна.
2. Arvin, A. M. (1996): Varicella-zoster virus: overview and clinical manifestations, Semin. Dermatol., 15:4-7.
3. Nymadawa P., Enkhsaikhan D., Loparev V. N., Bostik V., Tuul R., Demkin V. V. (2010): Genotypes of Varicella- Zoster Viruses Isolated in Mongolia. Abstract book of the International Conference on Emerging Infectious Diseases, p. 96.
4. Д.Энхсайхан, В.Н. Лопарёв, В. Бостик, Р. Туул, Б.Дарма, В.В. Демкин, П. Нимадава (2010): Генотипирование вирусов Варицелла-Зостер, выделенных на территории Монголии. Вопросы вирусологии. 55(5) : 40-3.
5. Loparev, V., Rubtsova, E., Bostik, V., Govil, D.,Birch, C. J., Druce, J. D., Scmid, D.S., Croxson, M. (2007): Identification of five major and two minor genotypes of varicella-zoster virus strains: a practical two-amplicon approach used to genotype clinical isolates in Australia and New Zealand, J. Virol., 81:12758-12765.
6. Loparev, V. N., Gonzalez, A., Deleon-Carnes, M., Tipples, G., Fickenscher, H., Torfason, E. G., Schmid, D. S. (2004): Global identification of three major genotypes of varicella-zoster virus: longitudinal clustering and strategies for genotyping, J. Virol., 78 : 8349-8358.
7. Robert D. Barber , Dan W. Harmer , Robert A. Coleman and Brian J. Clark (2005): GAPDH as a housekeeping gene: analysis of GAPDH mRNA expression in a panel of 72 human tissues. Physiological Genomics, 21:389-395).
8. Loparev, V. N., Argaw, T., Krause, P. R., Takayama, M., Schmid, D. S. (2000): Inmproved identification and differentiation of varicella-zoster virus (VZV) wild-type strains and an attenuated varicella vaccine strain using a VZV open reading frame 62-based PCR, J. Clin. Microbiol., 38:3156-3160.
9. Loparev, V. N., McCaustland, K., Holloway, B.P., Krause, P. R., Takayama, M., Schmid, D. S. (2000b): Rapid genotyping of varicella-zoster virus vaccine and wild-type strains with fluorophore-labeled hybridization probes. J. Clin. Microbiol., 38: 4315-4319.
10. La Russa,P., Lungu, O., Hardy, I.,Gershon, A., Steinberg, S.P., Silverstein, S. (1992): Restriction fragment length polymorphism of polymerase chain reaction products from vaccine and wild-tipe varicella-zoster virus isolates, J. Virol., 66:1097-1100.
11. Loparev V.N., Rubtcova E.N., Bostik V., Tzaneva V., Sauerbrei A., Robo A., Sattler-Dornbacher E., Hanovcova I., Stepanova V., Splino M., Eremin V., Koskiniemi M., Vankova O.E., Schmid D.S. (2009): Distribution of varicella-zoster virus (VZV) wild-type genotypes in northern and southern Europe: evidence for high conservation of circulating genotypes. Virology, 383(2): 216-225.
12. Sergeev N, Rubtcova E, Chizikov V, Schmid D.S, Loparev V.N. (2006): New mosaic subgenotype of varicella-zoster virus in the USA: VZV detection and genotyping by oligonucleotide-microarray, J Virol Methods. 136(1-2):8-16.
13. Liu J, Wang M, Gan L, Yang S, Chen J. (1995): Genotyping of clinical varicella-zoster virus isolates collected in China. J Clin Microbiol, 2009. 47(5): p. 1418-23.
14. Kim K.H., Choi Y.J., Song K.H., Park W.B., Jeon J.H., Park S.W., Kim H.B., Kim N.J., Oh M.D. (2011): Genotype of Varicella-Zoster Virus Isolates in South Korea. J Clin Microbiol. Epub ahead of print.
15. Loparev, V. N., Martro, E.,Rubtsova, E.,Rodrigo, C., Piette, J.C., Caumes, E., Vernant, J. P., Schmid, D.S., Fillet, A.M. (2007b): Toward universal varicella-zoster virus genotyping: diversity of VZV strains from France and Spain, J. Clin. Microbiol., 45:559-563.
16. LaRussa P., Steiberg S., Arvin A., Dwyer D., Burgess M., Menegus M., Rekryt K., Yamanishi K., Gershon A. (1998): Polymerase chain reaction and restriction fragment length polymorphism analysis of varicella-zoster virus, isolated from United States and other parts of the world. J. Infect. Dis. 178 (Suppl. 1): 64-66.




