1Эрүүл Мэндийн Шинжлэх Ухааны Их сургууль
2Анагаах Ухааны Хүрээлэн
Хүний биеийн өндөр нуруулаг галбир олон хүчин зүйлээс хамаардаг зүстөрхийн шинж юм. Эхийн эрүүл мэнд, нийгэм эдийн засаг, орчны хүчин зүйл, хоол тэжээл, өвчлөл, биеийн өндөрт нөлөөлөхөөс гадна сүүлийн жилүүдэд хүн амын дундаж өндрийг нэмэгдүүлсэн акцелерацийн үзэгдэл дэлхийн олон оронд нийтлэг ажиглагдах болсон билээ. Хэдий тийм боловч удам төрлийн олон үеийг хамруулсан ургийн модны судалгаа, олон ихрүүдийг оролцуулсан удмын судалгаанаас үзэхэд насанд хүрсэн хүмүүст өндөр чацархуу шинж өвлөгдөх байдал бараг 80-90% анхдагчаар удамшлаас хамаардаг байна (1,2,3). Биеийн өндрийн хэмжээс нь хүн амын дотор хэвийн тархалттай байдаг, хэмжилт хийхэд хялбар, насан турш тогтвортой хадгалагддаг, хүүхдүүд нь том болоод хир зэрэг нуруулаг болохыг эцэг эхийн биеийн өндрийн нийлбэрийн дунджаас тооцоолоод бараг ташаагүй үнэн хэлж болдог, удамшихуй ба регрессийн ухагдахуунд багтдаг зэргээс биеийн өндрийн судалгаа түүхэн үеийн турш генетикчдийн анхаарлын төвд оршсоор иржээ (4,5).
Анх Фишер Р.А. “Менделийн хуулийг батлаж буй ураг төрлийн хоорондын корреляци” бүтээлдээ өндөр хүмүүсийн төрхийг удамшлын олон хүчин зүйлс тус тусдаа удмаас удамд дамжуулдаг тухай санаа дэвшүүлсэн байдаг (6). Тэр цагаас хүний биеийн өндөр нь олон ген эвсэн өвлүүлдэг полиген хэвшинжийн сонгомол төрх болохыг бараг зуу гаруй жил судалсан боловч түүнийг хариуцдаг ген, геномын хэсгүүд, түүний вариацыг нөхцөлдүүлдэг молекулын механизмыг бүрэн нээн илрүүлж чадаагүй байна.
Биеийн өндөр нь зарим төрлийн хавдар, булчирхайн өвчин, хүүхдийн өвчин болон хүний бусад олон төрлийн хүнд нийлмэл өвчнүүдийн эрсдэлт хүчин зүйл болдог учраас түүний удамшлын механизмын тайлалаас гарах анагаах ухааны болоод биологи, танин мэдэхүйн хүлээгдэж буй үр дүн хэмжээлшгүй их юм (7,8).
Зарим төрлийн мутац эсвэл хэт өндөр, эсвэл хэт давжаа, одой, эсвэл бие цогцсын хүнд хэлбэрийн зэрэмдэг согогоор илэрдэг нь эрт дээр цагаас мэдэгдэж байсан авч ийм төрлийн гэр бүлийн мутац хүн амын хэмжээнд аваад үзэхээр нэн ховор тохиолдох ба өндрийн вариацын удамшлын үндсийн өчүүхэн бага хэсгийг тайлбарлах боломж олгодог.
Биеийн өндөр бол удамшлаар нөхцөлддөг үл маргах загвар төрх мөн боловч түүний материаллаг үндэс, молекулын механизмыг тогтоох нь амаргүй зорилт байсан, одоо ч байсаар байгааг олон сурвалж бичигт тэмдэглэн үлдээжээ. Генийн сан хөмрөгийн хамааралт хэлхээсийг гүйлгэн хайх (Genome–wide linkage scan) аргаар судлахад гарцаагүй өндөртэй хамааралтай ноёлол цэгүүд олноор олддог боловч тэдгээр цэгт байрлах өвөрмөц ген, генүүдийг дүйн тодорхойлох ажил ямагт амжилтгүй төгсдөг (1,9,10). Тэр нь нэг талаас сул нөлөөллийн нийтлэг аллель илрүүлэхэд хамааралт хэлхээсийн арга судалгааны хүчирхэг хэрэгсэл болж өгч чаддаггүйтэй холбоотой юм (11,12,13). Хүн амын томоохон бүлгийг хамруулсан судалгаанд биеийн өндөртэй хамааралтай төлөөлөл ген, генийн бүсүүд бэлхнээ нотлогдоод байхад уг судалгаанд ашигласан хамааралт хэлхээсийн арга ба ДНХ-ийн дэслэл давтан тогтоох (DNA resequencing) аргын аль аль нь өвөрмөц ген дүйн тодорхойлох зорилтыг шийдвэрлэхэд нэмэр болдоггүй байна(14,15,16).
Хүний биеийн өндөр бол хүн амын бүлэг тус бүрт өвлөгдөх индекс нэн өндөр (h2 ~0.8~0.9) шинж төрх боловч одоогоор мэдэгдээд байгаа ба одоогоор нээгдээгүй байгаа удмын олон хүчин зүйлийн хослолоор нөхцөлддөг полиген хэвшинж учраас түүнийг судлахын тулд саяхныг болтол хэрэглэж ирсэн анализын аргууд, технологийн түвшин, математик загварчлалын аргууд төгс биш байснаас түүний нууцад нэвтрэх оролдлого бараг 2007 он хүртэл амжилт олоогүй юм.
Харин “Хүний Геномын Төсөл” (Human Genome Project) болон уг төслийн хүрээнд санаачлан хэрэгжүүлсэн дэд төсөл болох “Дантөрхийн Зурагзүйн Консорциум” (HapMap Consortium)-ын гаргасан хаплотипын нэг нуклеотидын полиморфизм (ННП, англ. SNP-Single nucleotide polymorphism)-ийн иж бүрэн зураглалыг хийж гүйцэтгэх явцад зохион бүтээсэн технологийн дэвшил, тооцооллын загвар хүний генетикийн салбарт эргэлт авч ирсний ачаар биеийн хэмжээст зүстөрхийн (Quantitative Traits Loci) нууцад нэвтрэх үүд хаалга нээгдсэн юм. HapMap Consortium-ын ашигласан ННП(SNP), Дантөрх (Haplotype), Зүүлт ННП (tag SNP) зэрэг ухагдахууны Nature сэтгүүлд нийтлэгдсэн тодорхойлолтыг тойм байдлаар зураг 1-д дүрслэн үзүүлэв.
Энгийн байдлаар жишээлэхэд А/G гэсэн хоёр аллель бүхий ННП (SNP) намхан эсвэл өндөр хүмүүсийн аль бүлэгт олон тохиолдож байгааг судлахын тулд урьд нь ДНХ-ийн бүрэн хэмжээний дэслэлийг хүн бүхэн дээр давтан тогтоож уг ННП-ийг олох хэрэг гардаг байв(17,18). Геномын хөмрөгийг нэлэнхүйд нь хамарсан энэ төрлийн генотайпингийн судалгаа үлэмж өндөр өртөгтэй бөгөөд амьдрал дээр хэрэгжүүлэх боломжгүй юм. Энэ талаасаа Хүний Геномын Төсөл (19,20), Дантөрхийн Зураглалын Төсөл (21,22)-ийг амжилттай хэрэгжүүлсэн нь түүхэн ач холбогдолтой үйл явдал болсон бөгөөд өнөөгийн нөхцөлд зүүлт ННП (tag SNP) дээр үндэслэсэн генотайпингийн
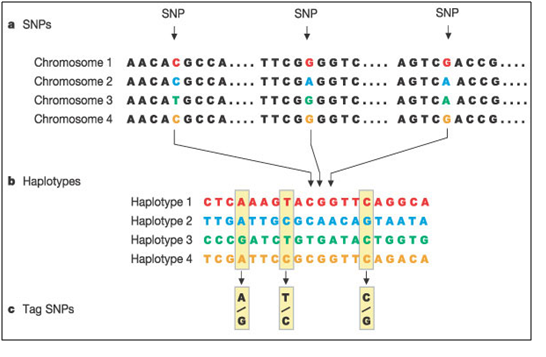
Зураг 1 ННП, Дантөрх, Зүүлт ННП.
a. ННП (SNP). Дөрвөн хүний нэг ижил хромосомын ДНХ-ийн яг ижил хэсгийг харьцуулахад ихэнх дэслэл адилхан, зөвхөн гурван цэг дээрхи нуклеотид (азотот суурь) ялгаатай байна. Нуклеотид бүр хос аллельтэй байдаг болохоор нэгдэх нуклеотидын полиморфизм эсвэл C эсвэл Т байх ёстой. b. Дантөрх(Haplotype). Дантөрх бол зэргэлдээх ННП-ийн хослол. ДНХ-ийн 6000 суурийн дагуу хайлт хийхэд a. хэсэгт үзүүлсэн гурван нуклеотидын вариантыг оруулаад бүгд 20 цэг дээр ялгаатай ННП-ийн генотип олдсон байна (зөвхөн полиморф хэлбэршилтэй нуклеотидыг сонгож аваад дөрвөн өөр өнгөөр харуулжээ). Haplotype 1-4 бол хүн амын дотор хромосомын энэ хэсэгт байж болох дантөрхийн дөрвөн хувилбар юм. c. Зүүлт ННП(Tag SNPs). 20 ННП-ээс зөвхөн гурван зүүлт ННП-ийн генотайпинг хийхэд дөрвөн дантөрхийг ялган дүйх бүрэн боломжтой юм. Жишээ нь, тухайн хромосом гурван зүүлт ННП дээрхи А-Т-С аллельтэй байвал энэ полиморфизм дантөрх 1-ийг тодорхойлно (хүн амд олон хромосом нийтлэг дантөрхтэй байдгийг анхаарах хэрэгтэй).
бүтээц (platform) ашиглан нэг ДНХ-ийн сорьцонд 1 сая орчим ННП-ийн хайлтыг хямд өртгөөр, алдаа мадаггүй зөв тодорхойлж (23,24) геномын хөмрөгийн бүх хэсгийг үлдээлгүй нэгжлэг хийж хайх боломж нэгэнт бүрджээ. Учир нь, геном дахь удмын вариант хоорондын корреляцыг нөхцөлдүүлдэг тэнцвэр алдагдсан хамаарал (TAX, англ. LD-Linkage Disequilibrium)-ын зохицлоор эдгээр нэг сая орчим ННП-ийг ашиглаад хүний геномын хөмрөг дотор 10 сая ННП-ийг дүйн тодорхойлж болдог байна (25). Тийнхүү генийн сан хөмрөгийг бүхэлд нь хамруулж хайлт хийх нүсэр ажлыг хөнгөвчилсний үр дүнд Геномын Хөмрөгийн Уялдаа (ГХУ, англ.GWA-Genome-Wide Association)-ны судалгаа үлэмж урагшилж хүний олон төрлийн өвчлөлтэй уялдаатай болох нь давтан батлагдсан ген, генүүдийн нийтлэг вариант олныг дүйн тодорхойлсон бөгөөд одоо үргэлжлэн тодорхойлсоор буйгаас гадна хүний биеийн өндөр, биеийн жин, липидын хэмжээ зэрэг биеийн тоон хэмжээст зүстөрхийг (ТХЗ, англ. QTI - Quantitative Traits Locus) хариуцдаг удмын үндсийг дүйн тодорхойлох ажил ихээхэн хурдацтай урагшлах болов. ГХУ судлалын нүсэр ажлыг хөнгөвчилж хялбар болгохын сацуу хуурамч эерэг нотолгоотой тулгарахаас сэргийлж чадахуйц чанд нягт боловсруулсан аргазүйн цэгнүүр мөрдөж ажиллах шаардлага гардаг (26,27). Үүнд, нэгдүгээрт, маш олон уялдааны сорил нэгэн зэрэг хийхэд статистикийн магадлал тооцох Р утгын босго 5 х 10-8 –ээс бага хүртэл өндөр нарийвчлалтай байх ёстой (28) бөгөөд үр дүнг олон тооны үл хамааралт хуулбарлалын (independent replication) шинжилгээгээр баталгаажуулах шаардлагатай. Хоёрдугаарт, уялдааны үзүүлэх нөлөө бага нөхцөлд хангалттай статистикийн хүч бүхий үнэн магадтай үр дүн гаргаж авахын тулд генотайпингийн шинжилгээг ДНХ-ийн маш олон сорьцонд хийх хэрэгтэй. Гуравдугаарт, генотайпингийн шинжилгээний болзошгүй техникийн алдаанаас сэргийлэх чанарын хяналтын зохистой цогц арга хэмжээ авах шаардлагатай (29) ба хүн амын ангиллыг зөв хийх, ялангуяа удам угсааны өөр өөр угшил гаралтай хүний генотайпинг уялдааны судалгаанд суурь нөлөө үзүүлдэг (30,31,32) тул зөв бүлэглэх асуудал их чухал. Эцэст нь, уялдааны судалгаанаас гарах асар их өгөгдлийн утгыг үр дүнтэй задлан шинжлэх хүчирхэг программ хангамж хэрэгтэй (33,34). Чухам эдгээр онцлог цэгнүүрийн тохироо 2007 онд бүрдсэн учраас биеийн өндрийн ГХУ-ны судалгаа сүүлийн хэдхэн жилийн дотор нэн хурдацтай хөгжиж эхэлсэн юм.
2.2.2. HMGA2 ген ба GDF5 генийг нээсэн нь
Олон улсын Диабетын Удамзүйн Санаачлага (ДУС, англ. DGI-Diabetes Genetics Initiative) байгууллагаас Диабетын хэвшинж-2 (ДХШ2, англ. Т2D-Type 2 Diabetes)-ийн эрсдэлт хүчин зүйлсийг тогтоох зорилгоор 1500 орчим өвчтөнд 350,000 гаруй нэг ННП ашиглан генотайпинг хийж хяналтын бүлэгт авсан Скандинавын 1500 орчим хүний генотайпингийн дүнтэй солбицуулан харьцуулсан байна (35). Учир нь, судалгаанд хамруулсан эдгээр 3000 оролцогчийн биеийн өндрийн хэмжээ хадгалагдаж байсан тул ДУС-ын хамтлаг биеийн өндрийн ГХУ-ны судалгааг хамтруулан хийжээ (35). Энэхүү судалгааны үр дүнд гарсан хариуны өгөгдлүүдэд хийсэн анхан шатны анализ биеийн нуруулаг галбирыг нөхцөлдүүлдэг ген бүхий удмын шинж байж болох талаар дэвшүүлсэн хэд хэдэн таамналыг батлаагүй бөгөөд биеийн өндрийг хариуцах магадлалтай генийн локус олоогүй юм(Зураг 2a). Энэ судалгаатай нэгэн зэрэг Веллком Трастын Тохиолдол Хяналтын Консорциум (ВТТХК, англ. Wellcome Trust Case Control Consortium)-ын нэг салбар болох Нэгдсэн Вант Улсын Диабетын хэвшинж 2 (НВУДХШ2, англ. UKT2D-United Kingdom Type-2 Diabetes) салбар өөрийн хяналтанд байсан ДХШ2 оноштой 2000 орчим өвчтөнд биеийн өндрийн ГХУ-ны судалгаа бас хийжээ (28,36). ВТТХК-ын НВУДХШ2 салбарын судлаачдын хичээл зүтгэл бас талаар өнгөрч ДУС-ын эрдэмтдийн адил биеийн өндрийн генийн локус олоогүй байна. Гэвч хоёр групп судалгааны дүнгээ нийлүүлэн хавсарсан анализ хийж үзтэл 12-р хромосом дээр байдаг HMGA2 ген (high mobility group A2 – монг. НХБА2 - нэмүү хөдөлгөөнт бүлэг-А2)-ийн 3’ үл хөрвөх бүсэд байрлах ННП-ийн P утга 4x10-8 нарийвчлал бүхий магадлалтай (P<4x10-8) бусдаасаа тасархай хүчээр биеийн өндөртэй уялдаатай байжээ (Зураг 2b). Энэхүү HMGA2 ген ба өндрийн вариацын хоорондын уялдаа насанд хүрэгсдийн 20,000 хуулбарлалт (replication), хүүхдийн 6800 хуулбарлалтад тогтвортой хадгалагдаж биеийн өндөр ба ерөнхий ДНХ-ийн дэслэлийн вариацын хооронд магадтай уялдаа байдгийг хүлээн зөвшөөрөхөөс аргагүй байдалд хүргэсэн анхны нотолгоо болсон юм (37). Урьдчилан таамаглаж байсанчлан уг ННП-ийн биеийн өндөрт үзүүлэх нөлөөний хүч сул буюу С аллель бүхэн нь өндрийг 0,4 см-ээр нэмэгдүүлж байсан ба хүн амын бүлэгт биеийн өндрийн вариацын ердөө 0,3%-ийг нөхцөлдүүлж байжээ.
HMGA2 ген (Зураг 3) бол хуулбарлалтын идэвхтэй үүрэггүй хроматины уургийн товчилборыг хадгалдаг онкоген хэдий ч биеийн өндрийг хариуцдаг генийн сонгомол төлөөлөл болох нь дараах үндэслэлээр батлагдаж байгаа юм(38). Нэгдүгээрт, түүний хомологийн делец хулганд Пигми (Pygmy) мутантын зүстөрхийг нөхцөлдүүлдэг (39), хоёрдугаарт, HMGA2-ын тасархайтлын транслокацтай өвчтөнд хүнд хэлбэрийн хэт 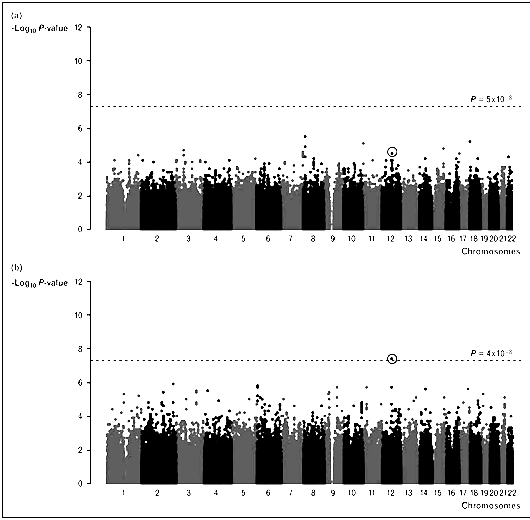
Зураг 2. ГХУ-ны судалгааны явцад нээсэн биеийн өндөр уялдаат HMGA2 ННП
(а) ДУС-ийн хамтлагийн хийсэн 3025 хүний генотайпингийн дүнг харуулсан Манхеттений толбон цамхаг. Босоо y-тэнхлэг дээр хамаарлын Р утга (логарифмын шатлалаар), хэвтээ x-тэнхлэг дээр бүх аутосомд хийсэн генотайпингийн 386 371 ННП-ийн Р утгын толбо (толбо болгон өөр өөр ННПХ-ийг заана). Хамааралтай байж болох магадлалын утганд (тасархай шугам, P < 5 x 10-8) ННП-ийн нэг ч Р утга дөхөж очоогүй байна. (b) ДУС ба ВТТХК-ын ДХШ2-ын хавсарсан ГХУ-ны судалгааны дүнг харуулсан Манхеттений толбон цамхаг (хавсарсан судалгаанд оролцогчийн тоо = 4951). Бодитой уялдаатай нэг толбо (P <4 x 10-8) магадлалын шугаманд хүрч байна (цагираг дотор). Энэ бол HMGA2 генийн 3’ үл хөрвөх бүсэд байрлах ННП-ийн толбо.
өндөрсөх хам шинж үүсдэг (40), эцэст нь HMGA2 локус алдагдсан микроделецтэй хүмүүс эмгэг хэт давжаа бие цогцтой болдог байн а(41). HMGA2 генийн тухай мэдээлэл нийтлэгдсэнээс тун удалгүй Финланд-АНУ-ын инсулин–үл хамааралт чихрийн шижингийн судалгааны хамтлаг (FUSION) ба Genetics, SardiNIA төслийн судлаачдын хамтарсан баг ГХУ-ны өндрийн судалгааны дүнгээ нэгтгэж 20 дугаар хромосом дээрхи GDF5 генийн ойролцоо орших биеийн өндөртэй уялдаатай хоёрдахь локусыг нээжээ (42).
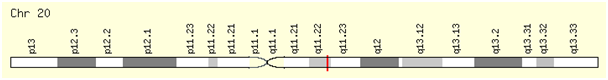
Зураг 3. HMGA2 gene (НХБА2 ген). Генийн нэршлийн хорооны (HGNC) дугаар: 5009 (генийн байрлалыг улаан зураасаар харуулав)
GDF5 ген (Зураг 4) бол нийт ДНХ-ийн олон вариантад сайтар нууцлагдсан биеийн өндрийн вариацын модулийн нэг мөчирлөл зохицуулгын чухал төлөөлөл юм (42). Учир нь, GDF5 (growth differentiation factor 5, монг. ӨЯФ5- өсөлт ялгарлын фактор-5) генийн ховор мутац ахар хурууны согог (англ. brachydactilia) болон мөгөөрсний дисплази (chondrodysplasia) үүсгэдэг (43,44) ба яг биеийн өндөртэй уялдсан ННП-ийн тэр хэсэг нь бас остеоартрит үүсэх эрсдэл агуулдаг ажээ (45).
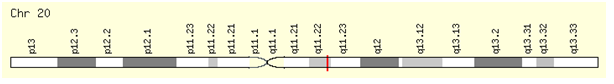
Зураг 4. GDF5 gene (ӨЯФ5 ген). Генийн нэршлийн хорооны (HGNC) дугаар: 4220 (генийн байрлалыг улаан зураасаар харуулав)
HMGA2 ген ба GDF5 ген оршдог локусын генетикийн вариац биеийн өсөлтөд хэрхэн нөлөөлдөг, болон тэдгээрийн молекулын механизмыг тайлах судалгаа өнөө хир эрчимтэй үргэлжилсээр байна. Түүний зэрэгцээ HMGA2 ба GDF5 генийн локусын нээлтээс үлгэр авсан судалгааны олон хамтлаг өндрийн нэмэлт локус илрүүлж дүйн тодорхойлох чиглэлийн хойноос хушуурч ГХУ-ны өндрийн судалгааны дүнгүүдээ нэгтгэн хавсарсан дам-анализ (meta-analysis) хийж богино хугацааны дотор олон шинэ ген нээсэн байна.
2.2.3. Биологийн үүрэг жамын ондоошил бүхий өндрийн ген, геномын дам-анализын судалгаа
2007-2008 оны зааг дээр Gudjbartson D.F, Lettre G, Weedon MN, Johansson A, Soranzo N, нар жил хүрэхгүй хугацаанд ИБУИНВУ, Скандинавын орнууд, Голланд арлын улсууд, Исландад эрт дээр цагаас оршин сууж ирсэн европын уугуул цагаан арьст кавказойд элэнц угшилтай 80,000 шахам хүн амын бүлгийг хамруулсан том хэмжээний дам-анализын хэд хэдэн судалгаа хийж гүйцэтгэсний дүнд биеийн өндрийн вариацтай холбоотой 50 гаруй ННП шинээр нээж баталгаажуулсан байна (46,47,48,49,50). Тухайлбал, Lettre G нар (2008), диабетын хоёр (DGI, FUSION), хавдрын хоёр (NHS-Nurses’ Health Study, монг.СЭМС-Сувилагчийн эрүүл мэндийн судалгаа, PLCO-Prostate, Lung, Colon and Ovarian Cancer Screening Trial, монг.ТУБГӨ-Түрүү, уушиг, бүдүүн гэдэс, өндгөвчийн хавдрын судалгаа), хүн ам зүйн хоёр (KORA-Германы өмнөд нутгийн Augsburg мужийн хүн амын удамзүйн судалгаа, SardiNIA-Италийн Сардинийн Ogliastra мужийн өндөр настны удамзүйн судалгаа), бүгд зургаан өөр төрлийн судалгаанд хамрагдсан нийт 15’821 оролцогчийн өгөгдлийг ашиглан биеийн өндрийн ГХУ-ны дам-анализ хийжээ (47). Оролцогчдын тайпингийн шинжилгээг Affimetrix 500K (DGI, KORA, SardiNIA), Illumina 317K (FUSION), Illumina 550K (NHS, PLCO) гэсэн өөр өөр бүтээц дээр тулгуурлан хийсэн учраас хооронд нь оноон тохируулах, ижилтгэн жигдрүүлэх зорилгоор МАСН программ (Y.Li, ба G.R.A) ашиглан HapMap төслийн хоёрдахь жишигтэй нийцсэн 2’260’683 ННП гаргаж авсан байна. Биеийн өндрийг эдгээр ННП-тэй уялдуулан тус бүрт нь удмын нэмэлт загвараар судалсан бөгөөд үр дүнг цэгнэгдсэн Z онооны арга ашиглан дам-анализаар нэгтгэжээ. Судалгааны зургаан бүлгийн уялдааны статистикийн тархалтыг тус тусд нь авч үзэхэд квантил-квантил талбай дээр Р утгын тархалт бага буюу бүгд уялдаагүй гарчээ (Зураг 5). Гэтэл зургаан бүлгийн 15’821 хүний тархалтыг нэгтгээд тооцоолоход 12 локус уялдаатай болох нь нотлогдсон байна. Үүнээс хамгийн өндөр уялдаатай гурван локусын хоёр нь буюу rs1042725 (P = 2,6 x 10-10) ба rs6060369 (P = 1,9 x 10-10) гэсэн хоёр локус нь өмнө нээгдсэн HMGA2 ген ба GDF5 генийн локусууд байжээ. Тийм болохоор уялдааны утгаараа энэ хоёр локустай ойролцоо буюу давж гарсан утгатай бусад локус лавтайяа уялдаатай байх магад гэж үзээд лавшруулан судалсаар биеийн өндөртэй уялдаатай 10 локус шинээр нээсэн байна (Зураг 6a).
Тэдгээр нь ихэвчлэн биологийн жамын хувьд санаанд оромгүй ондоо үүрэг ажилтай генийн бүсэд оршиж байв. Энэ өнцгөөс нь аваад үзэхээр биологийн үүрэг ажлын ондоошил бүхий энэ олон генийн дам-анализын судалгаа биеийн өсөлтийн биологийг
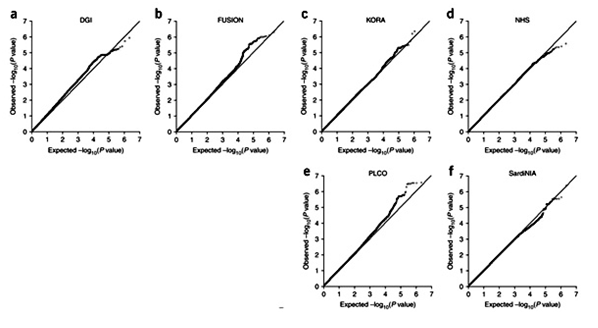
Зураг 5. Зургаан өөр төрлийн судалгаанд хамрагдсан оролцогч тус бүрийн ГХУ-ны дүнг тус тусд нь тооцсон дам-анализын ~2,2 сая гаруй ННП-ийн тархалтын квантил-квантил талбай.
(a) DGI (n = 2,978), (b) FUSION (n = 2,371), (c) KORA (n = 1,644), (d) NHS (n = 2,286), (e) PLCO (n = 2,244), (f) SardiNIA (n = 4,305). Хар толбо болгон оролцогч нэг бүрийн тооцсон статистикийн эсрэг тооцоолж гаргасан хос цувааны Р утгын сөрөг логарифм (log10). Шулуун шугам тэг тархалттай тохирно. Р утгын босго 5 х 10-8 –ээс давсан уялдаа нэг ч талбайд харагдахгүй байна.
танин мэдэх салбарт судалгааны асар олон шинэ чиглэл, өргөн хэмжээний судалгааны шинэ талбар нээж байгаа юм.
Шинээр нээсэн локусуудын дотроос ННП rs1492820 локус сэвлэг дохиол нэгдлийн уураг (СДНУ, англ. HHIP-Hedgehog-interacting protein)-ийн ген дотор олдсон бөгөөд энэ уураг сэвлэг дохиолын транскрипцийг саатуулах үүрэгтэй ба хөхтөнд гурван төрлийн сэвлэг дохиолын уурагтай өндөр авцаал (high avidity)–тай нэгддэг. Түүний хулганы хомолог Hhip уураг нь мөгөөрсний ирмэг зах (perichondrium)-аар болон цуваан сэвлэг дохиол(ЦСД, англ. Ihh-Indian hedgehog)-ын давхцаа бүсийн генийн бүтээдэг араг ясны нуруу нумын тууш болон дагуул хавиар илэрдэг. Hhip уургийн хулганы мөгөөрс дахь төөрүү хэт илрэл (ectopic overexpression) Ihh тэг хулганд илэрдэг зүстөрхийг санагдуулам богино мөчит одой болон бусад хүнд хэлбэрийн араг ясны согог үүсгэдэг байна. Хэд хэдэн ННП хроматины бүтэцтэй холбоотой ген дотор юмуу ойролцоо олдсон байна. Тухайлбал, хроматин холбогч уургийн товчилбор хадгалдаг HMGA2 – оос гадна хромосом 6 дээрхи хистоны цомог дотор ННП rs10946808, хистон метилтрансферазын DOT1L ген дотор ННП rs12986413,
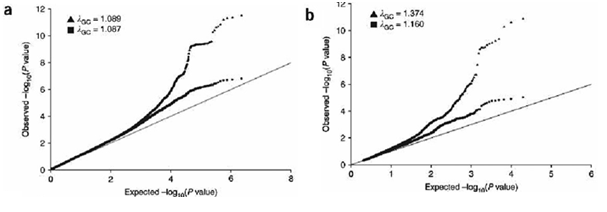
Зураг 6. Зургаан өөр төрлийн судалгаанд хамрагдсан оролцогч тус бүрийн ГХУ-ны дүнг нэгтгэн тооцоход илэрсэн биеийн өндөртэй уялдаатай локусуудыг харуулсан квантил-квантил талбай.
(a) 15,821 оролцогчийн ГХУ-ны дүнг нэгтгэсэн дам-анализын 2,260,683 ННП-ийн тархалтын квантил-квантил талбай. Хар гурвалжин болгон оролцогч нэг бүрийн тооцсон статистикийн эсрэг тооцож гарсан хос цувааны Р утгын сөрөг логарифм (log10). Хар дөрвөлжин болгон Р утгын босго 5 х 10-7 –ээс давсан уялдаа бүхий 12 локусыг хассаны дараа тархалт. Шулуун шугам тэг тархалттай тохирно. (b) Уялдааны утгааараа тэргүүлэх 10’000 ННП-ийн тархалтын квантил-квантил талбай (хар гурвалжин болгон биеийн өндөртэй уялдаатай 12 локусыг хасахын өмнө тархалт, хар дөрвөлжин болгон хассаны дараа тархалт).
метилжсэн ДНХ холбох транскрипцийн саатлуур ZBTB38 генийн интрон дотор ННП rs724016 олджээ. Хроматины архитектурын тогтоцын үүрэгтэй эдгээр ген биеийн галбир, зүстөрхийн тохируулгад ямар үүрэгтэй байж болох нь одоохондоо ойлгомжгүй байгаа юм. Гэхдээ алсуураа холбоотой байж болох нэг баримт байдаг. Биеийн галбир хэт өндөр шинж тэмдгээр илэрдэг Сотосын хамшинж (ОМИМ117550) хистон метилтрансферазын NSD1 генийн мутац болон делецээс болдог. Тийм болохоор HMGA2, хромосом 6 дээрхи хистоны цомог, DOT1L, ба ZBTB38 генүүд дээрхи өндрийн вариантууд Сотосын эмнэл зүйн шинж тэмдэг үүсэхэд хүргэдэг эсэх, эсвэл эдгээр ген ялангуяа DOT1L генийн хүнд хэлбэрийн мутац Сотос төст хамшинж үүсэхэд оролцдог эсэхийг тогтоох хэрэгтэй.
Насанд хүрэгсэд ба хүүхдийн биеийн өндөртэй хүчтэй уялдаатай rs1042725 вариант HMGA2-ийн 3’ үл хөрвөх бүсэд олдсон нь ихээхэн анхаарал татах үзэгдэл мөн. HMGA2 бол хүний бусад ген дотроос let-7 бичилРНХ (microRNA) таних залгуур сайт (binding site) хамгийн олныг агуулдаг онкоген. Яг байрлалын хувьд rs1042725, let-7 таних залгуур сайтаас 13 суурь зайтай оршдог учраас богочлогдож (splising) let-7 бичилРНХ хүчилтэй нэгдэх замаар HMGA2 ген илрэх механизмд оролцдог байж болох таамналд хүргэж байгаа юм.
Lettre G нарын нээсэн шинэ локусуудаас бас өөр гурван локус let-7 таньж залгагддаг эсийн мөчлөг тохируулгын CDK6 ген (cyclic-dependent kinaze-6, монг. МХК-6-мөчлөг хамааралт киназ-6), хистон метилтрансферазын DOT1L ген, LIN28B ген дотор олдсон байна. ННП rs7869550 бас л let-7 таних залгуур сайт агуулдаг байж болох PAPPA ген дотор олджээ. Тийм болохоор let-7 бичилРНХ хүчлийг өөрийг нь болон түүнтэй залгагддаг бай генүүдийн биеийн өндрийн тохируулгад оролцдог механизмыг бас судлууштай.
Шинээр олдсон G-уургийн рецепторын GPR126 ген (G-protein-coupled receptor 126, монг.ГУХР126-Г-уураг-хослох рецептор-126), бамбайн дааврын рецепторын зууч уургийн TRIP11 ген (thyroid hormone receptor interactor protein-11, монг.БДРЗУ-11-бамбайн дааврын рецепторын зууч уураг-11), атаксины ATXN3 ген (ataxin N3, монг.АТКСН3-Атаксин-Н3), Хантингтон-нэгдлийн SH3GL3 ген бүхий локус, гликопротейн металло-протеазын ADAMTS3 ген (энэ генийн мутац ихэвчлэн бүдүүн гэдэсний хавдар үүсгэнэ), шүлсний булчирхайн аденомын PLAG1 онкогентэй ямагт хамсах чанартай CHCHD7 ген (coiled-coil-helix-coiled-coil-helix domain containing-7, монг. МОМОХ7-мушгиа-ороомог, мушгиа-ороомог, хэрчим-7) бүхий локус, эпидермисийн эвэрлэгийн дехидрогеназын RDHE2 ген зэрэг локусууд нь биеийн өндөр болон галбир цогцтой ямарч холбоогүй генийн бүсийн ойролцоо олджээ.
Lettre G нарын энэ судалгаанд хүчтэй уялдаатай бүлэгт багтаагүй ч гэсэн уялдааны хүч арай сул бүлэгт орсон генүүд дотор анхаарал татахуйц ННП хэд хэд байгаа юм. Мөчлөгт гуанозин монофосфат хамааралт протейнкиназ II (cGKII, монг.мГКII-мөчлөгт Гуанозилкиназ II)-ийн толчилборыг хадгалдаг PRKG2 генийн өгсөх чигийн 28 kb-д олдсон rs662845 ННП бол биеийн өндрийн вариацд чухал үүрэгтэй локус байх ёстой. Учир нь, нэгдүгээрт, туршлагын амьтны загвар дээр Prkg2-/- хулганд мөгөөрс голоосоо ясжих процесс саатах хүнд хэлбэрийн одойн согог илэрдэг ба хоёрдугаарт, байгал дээр тохиолддог Ишикавагийн мутант Комедагийн бичил харханд cGKII генийн делецээс болсон биеийн тууш өсөлтийн хоцрогдол үүсдэг. Энэ нь харх болон мэрэгчдэд хондроцитын хуваагдал төгсмөгц ялгарал эхлэх молекулын шилжилтэд нөлөөлдөг cGKII ген араг ясны хөгжилд чухал үүрэгтэй болохыг нотлож байгаа юм. Энэ талаас нь аваад үзэхээр түүний хүний хомолог PRKG2 генийн биеийн өндрийн вариацийн оролцоог том хэмжээний хүн амын бүлэг оролцуулсан хуулбарлалтын судалгаагаар батлах боломжтой нь харагдаж байна.
Lettre нарын энэ судалгааны дүнд нээсэн хүчтэй уялдааны бүлгийн хэд хэдэн локус (ZBTB38, HMGA2, GDF5, HHIP, SH3GL3-ADAMTS3, CDK6), Weedon нарын (2008) шинээр тогтоосон локустай яв цав тохирч энэ хоёр судалгаа бие биеэ харилцан баталгаажуулсан байна. Үүний зэрэгцээ Weedon нарын судалгаанд биеийн өндөртэй хүчтэй уялдаатай байсан FUBP3 ген дотор олдсон ННП, Lettre нарын энэ судалгааны арай сул уялдааны бүлэг доторхи FUBP3 гентэй бас тохирч байжээ. Энэ FUBP3 ген c-myc тохируулгын үүрэгтэй учраас өндрийн вариацын бас нэг төлөөлөл байх бүрэн үндэстэй юм.
Lettre нар хатуу шалгуураар шигшиж авсан эдгээр 12 локусыг хасаад харахад квантил-квантил талбайн тэг тархалтын шулуунаас хэвийж алсран холдох Р утгын хандлага өөрчлөгдөөгүй байна (Зураг 6a, хар дөрвөлжин). Түүгээр зогсохгүй хүчтэй, арай сул, сул уялдаатай бүх локусыг хасаад юу үлдэхийг харах зорилгоор тэргүүлэх уялдаатай 10’000 ННП-ийг хасаад харахад уялдааны Р утга тэг тархалтаас холдсон хэвээр байжээ (Зураг 6b, хар дөрвөлжин). Эндээс харахад биеийн өндөртэй уялдаатай бусад ген, геномын хэсгүүдийг бүрэн нээж дуусах болоогүй, цаана нь шинээр нээх ёстой ген олон байгаа, хүн амын илүү олон бүлгийг хамруулсан өргөн цар хүрээтэй судалгаа хийх шаардлагатай болох нь харагдаж байгаа юм. Энэ зорилгоор олон улсын хэмжээнд “Антропометрийн Хэмжээсийн Удамзүйн Судалгааны Консорциум” (GIANTC - Genetic Investigation of ANthropometric Traits Consortium) байгуулж 120’000-аас доошгүй хүн оролцуулсан дам-анализ хийх нь зүйтэй гэж үзэх болов.
Биеийн өндөр уялдаат генийн хайлтын судалгааны тэргүүн шугам дээр өрсөлдөж гарч ирсэн бусад олон судлаачдын дотроос M.N.Weedon нар (2008), Gudbjartsson D.F. нар (2008)-ын судалгаа онцгой анхаарал анхаарал татдаг. M.N.Weedon нар ВТТХК-ын ДХШ2 (WTCCC-T2D) салбар, Даралт ихсэл судлах (WTCCC-HT-hypertension) салбар, Титэм судасны өвчин судлах (WTCCC-CAD-coronary artery disease) салбар зэрэг дөрвөн судалгааны бүлэг ба Цусны Үндэсний Төв (WTCCC-UKBS-United Kingdom Blood Service)-ийн бүлэг, Таргалалтын тохиолдол-когорт (Obesity case-cogort study) судалгааны бүлгийн нийт 16’482 хүнд хийсэн генотайпингийн дам-анализын дүнд 20 ген, Gudbjartsson D.F. нар 25’174 Исланд, 2876 Дани, 1770 Европын Америк, 1148 Африкийн Америк хүнд хийсэн генотайпингийн дүнгээ өмнө хийсэн 8541 хүний дүнтэй нийлүүлж хийсэн хавсарсан дам-анализын дүнд 27 ген тус тус шинээр олсон байна.
Эдгээр судлаачдын шинээр нээсэн ННП-ийн давхардсан генийг нэгтгээд хромосомын байршил, хувьсамжийн үүдэлтэй бүртгэгдсэн өвчин, үүрэг ажлын ондоошилтой уялдуулан хүснэгт-1-д хавсаргав.
Хүснэгт 1. Насанд хүрэгсдийн бие цогцтой уялдаатай хромосомын бүс ба төлөөлөл ген
.png)
Хүснэгтээс харахад одоогоор нээгдээд байгаа эдгээр ННП-ийн аллель бүрийн биеийн өндөрт үзүүлэх нөлөө бага буюу 0.2-0.5 см-ээс ихгүй бөгөөд нийт ННП нэгдсэн дүнгээрээ биеийн өндрийн зүстөрхийн вариацын 5 орчим хувийг нөхцөлдүүлж байна. Энэ нь биеийн өндрийн өвлөгдөх шинжийн хүчтэй нөлөөллийн бусад олон локус нууцлаг оршсоор байгааг харуулж байгаа юм (51).
Эдгээр локусын бүсэд орших генүүдээс олон тооны ген тэнцвэр алдагдсан хамаарлаар (ТАХ) олдсон тул цаашдын шалтгаан үрийн нарийвчилсан туршилтгүйгээр чухам аль ген буюу геномын хэсгүүд биеийн өндрийг гардан хариуцдагийг оноож тогтооход түвэгтэй байгаа юм.
Gudjbartson D.F нар (2008) одоогоор нээгдээд буй биеийн өндрийн ген, генүүдийн төлөөллийг үүрэг ажлын гурван бүлэг, тухайлбал, араг ясны хөгжилтэй холбоотой ген (BMP2, BMP6)-ийн бүлэг, мөгөөрсний бүтцийн өөрчлөлтийг тохируулдаг цайр хамааралт металлопротейназууд (ADAMTS10) ба гликопротеинуудын ген (FBN1)-ийн бүлэг, хромосомын сегрегаци, митозын процесст оролцдог ген (CDK6, HMGA)-ийн бүлэг гэж ангилах санал дэвшүүлсэн (46) бол Lettre G. нар (2008) хроматины бүтцэд оролцдог ген (HMGA1, HMGA2, DOT1L, хос гистоны цомог, SCMH1)-ийн бүлэг, яс мөгөөрсний эсийн гадна матриксийн уургуудыг бүтээдэг ген (ACAN, FBLN5, EFEMP1, ADAMTS17, ADAMTSL3)-ийн бүлэг, ясны морфогенезийн уургуудын дохиоллын ген (NOG, GDF5, BMP2, BMP6)-ийн бүлэг, эсийн мөчлөгийн тохируулгын ген (CDK6, CABLES1, ANAPC13, NCAPS)-ийн бүлэг, сэвлэг дохиолын замын ген (IHH, HHIP, PTCH1) -ийн бүлэг гэсэн таван бүлэгт хуваан авч үзэх санал дэвшүүлжээ (56). Үүний зэрэгцээ эдгээр бүлгүүдэд аль алинд нь хамаардаг нийтлэг ген, генүүд байдгийн нэг томруун төлөөлөл бол ZBTB38 ген юм. Энэ генийн цайран үдээст уураг (англ.zinc-finger protein) нь метилжсэн ДНХ, ялангуяа Н19/IGF2 (IGF2-Insulin-like growth factor 2, монг. ИТӨФ2- Инсулин-төст өсөлтийн фактор 2)-ын ялгарч метилжсэн бүсийн метилжсэн аллелльтэй сонгон нэгдэж хуулбарлалтыг зогсоодог (57). ДНХ-ийн энэ сайтын генетикийн дээхнэ алдаа (epigenetic errors) эсвэл хүний удамшлын Менделийн хуулийн цахим бүртгэл (ХУМХЦБ, англ. OMIM-Оnline Mendelian Inheritance in Man)-ийн 130650 дугаартай Беквит-Видерманы хэт өсөлтийн хамшинж (OMIM 130650), эсвэл, Руссел-Силверын хэт давжаа хам шинж (OMIM 180860) үүсгэдэг(55). Эндээс ургуулаад бодоход ZBTB38 ген хөврөлийн хөгжлийн шатанд өсөлтөд чухал үүрэгтэй ИТӨФ2-ын шүүрэлд нөлөөлөх замаар биеийн өсөлтийг тохируулах бүрэн үндэстэй байна(48). ZBTB38 ген метилжсэн ДНХ-д нөлөөлөхөөс гадна катехоламины нийлэгшлийг үечлэн хязгаарладаг тирозин хидролазын нууцалбар хадгалдаг ТН генийн хуулбарлалтыг бас хариуцдаг(47).
Түүнээс гадна бичилРНХ (microRNAs) хүчлүүдийн биеийн өндрийг зохицуулах хуулбарлалын дараа түвшний тохируулга (ХДТТ, англ. Posttranscriptional regulation) ихээхэн сонирхол татах болов (56). Учир нь биеийн өндрийг хариуцдаг олон ген (HMGA2, CDK6, DOT1L, LIN28B, PAPPA) ба генүүд let-7 бичилРНХ хүчлийн бай болж өгдөг.
Lettre G нарын (2009) европ угшилтай насанд хүрэгчдэд дүйн тодорхойлсон биеийн өндөртэй уялдаатай генийн 10 локусын 4 нь Weedon M.N. нарын (2008) нээсэн let-7 бичилРНХ-ийн нөлөөнд оршдог HHIP генийн дөрвөн локустай яг тохирч байжээ.
Эдгээр судлаачид HMGA2 ген, хистон метилтрансферазын DOT1L ген, метил-ДНХ-холбогч хуулбарлалтын хориг ZBTB38 ген гэсэн энэ гурван төлөөлөл ген хроматиныг давтан загваржуулахад оролцдог ба HMGA2 генийн 3’ үл хөрвөх бүсэд let-7 бичилРНХ хүчилтэй нэгддэг олон тооны залгуур сайтууд, мөн элэгний эсийн карциномын (hepatocellular carcinoma) ургалтыг дэврээдэг CDK6, DOT1L, LIN28 гэсэн бусад гурван ген нь бас let-7 хүчлийн үйлчлэл дор оршдогийг цохон тэмдэглэсэн байна. Ер нь энэхүү let-7 зэрэг жижиг хэмжээст үл хөрвөх РНХ хүчлүүд нөлөөндөө орших бай генүүдийн илрэлийг ямагт саатуулж байдаг ажээ.
ХДТТ-ын бас нэг сонгомол жишээ бол 7-р хромосом дээр байрлах биеийн өндөр ба ДХШ2 уялдаат YAZF1 ген юм(52). Энэ бол хуулбарлал саатуулагч ген бөгөөд биеийн өндөр ба ДХШ2 өвчинтэй уялдаагүй, корреляцын холбоосгүй дан хэлбэрээр оршихдоо түрүү булчирхайн хавдрын эрсдэлийг нэмэгдүүлдэг байна (53). Эдгээр ажиглалтууд хүний биеийн өндөр ба хавдар болон бусад өвчнүүдийн хоорондын зарим хамаарлыг тайлбарлах суурь болж өгч байгаа юм.
Дам-анализын судалгааны дүнд нээсэн ГХУ-ын олон ген, генүүд биеийн өндрийг хариуцахын зэрэгцээ араг ясны хөгжлийн хүнд хэлбэрийн алдагдал, мутац, бусад хэлбэрийн хувьсамжийн үндэс болж өгдөг байна. Тэдгээрийн дотроос 12, 20-р хромосом дээр байрлах HMGA2, GDF5 генээс гадна 15q26, 18q21, 2q35, 17q23, 9q22, 8q21, 17q11, 17q23 дээр оршдог ACAN, DYM, IHH, NOG, PTCH1, PXMP3, RNF135, TBX4 генүүд биеийн өндөрт нөлөөлөхөөс гадна зүстөрхийн гүнзгий өөрчлөлтөөр илэрдэг ховор тохиолдох гэр бүлийн мутацийг төрүүлдэг байна (Хүснэгт 1). Энэ нь ГХУ-ны судалгааны явцад нээгдээд байгаа өндрийн генүүд эсвэл өндөр галбир, эсвэл давжаа галбир, эсвэл араг ясны хөгжлийн согогоор илэрдэг хүний өнчин хамшинжүүд (англ. Orphan human syndromes) буюу шалтгаан үрийн уг үндэс болсон ген нь олдоогүй байгаа хамшинжүүдийг судлах хосгүй шинэ боломж олгож байна. Жишээ нь, бамбайн даавар молекултай харилцан үйлчлэлцдэг уураг 11-ийн генийн (TRIP11) ойролцоо орших нийтлэг ННП, ГХУ-ны судалгаагаар биеийн өндөртэй уялдаатай байжээ (46,47). Энэ тухай нийтлэл гарсны дараа, уг судалгаатай огт холбоогүй өөр нэг судалгаанд TRIP11 генийн бүтцэд гэмтэл учруулсан мутац хулгана болон хүний амь насанд халтай араг ясны үхлийн дисплази (lethal skeletal dysplasia) үүсгэдэг нь тогтоогдсон байна (54). Энэ нь шалтгаан тодорхойгүй өсөлтийн эмгэг болон араг ясны эмгэгтэй өвчтөнүүдэд өндрийн бусад генийн дэслэлийг тогтоох шинжилгээ нэвтрүүлбэл тухайн эмгэгүүдийн шалтгааныг молекулын этиологийн түвшинд оношлож болох тийм эрин цаг үе нэгэнт ирснийг харуулж байна. Түүнээс гадна одоогоор нээгдээд буй ННП-ийг ашиглаад өндөр намхан хүмүүсийг ангилах болов. Тухайлбал, томоохон хэмжээний хүн амын бүлэг хамруулсан Финландын когорт судалгаанд өндрийн 12 биаллельт ННП-ийн генотайпинг хийж үзэхэд биеийн өндрийг нөхцөлдүүлдэг 8 ННП агуулсан хүн 16 ННП агуулсан хүнээс 3,5 см-ээр намхан (47) байжээ (Зураг 7).
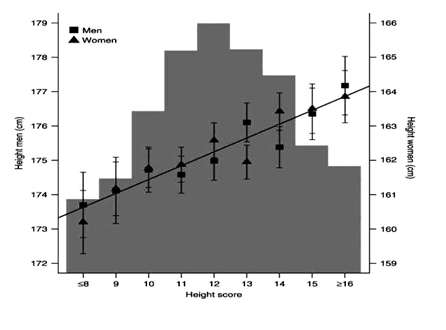
Зураг 7. Өндрийн ННП ба өндрийн зааглал
FINRISK97 когорт судалгаанд хамруулсан өндрийн 12 ННП-ийн генотип бүхий оролцогч нэг бүрийн (n=7566) аллелийн тоон дээр үндэслэн гаргасан өндрийн оноо (height score). Оролцогчдийг 8 хүртэл буюу түүнээс доош, эсвэл 16 хүртэл буюу түүнээс доош аллель агуулсан байдлаар бүлэглээд өндрийн онооны (12 би-аллелт ННП; өндрийн оноо 0-24 хооронд хэлбэлзэнэ) бүлэг тус бүрийг +/- 95%-ийн завсартайгаар эрэгтэй, эмэгтэй тус тусад нь тэмдэглэсэн байна. Босоо тэнхлэгийн баруун зүүн талд эрэгтэй, эмэгтэй бүлгийн өндрийн хэмжээг адил завсартай байрлуулжээ. Регрессийн шугам хоёр хүйсийн аль алинд хамаарах ба өндрийн аллель нэгээр нэмэгдэхэд биеийн өндөр 0,4 см-ээр нэмэгдсэн байна. 8 ННП агуулсан хүн 16 ННП агуулсан хүнээс 3,5 см-ээр намхан байна.
Хүний биеийн өндрийн өвлөгдөх шинжийн нээгдээгүй байгаа бусад ННП-ийг илрүүлэхийн тулд илүү олон хүн амын бүлэг, илүү олон угсаатны бүлэг хамруулсан судалгаа хийх шаардлагатай байгаа юм. Гэхдээ биеийн өндөр болон биеийн бусад хэмжээст зүстөрхийн ГХУ-ны судалгаа нийтдээ 600 гаруй уялдаат ННП (вариант) тогтоогоод байгаа хэдий ч нөлөөний хүч нь бага, судалж буй хэмжээсийн 10 орчим хувиас хэтрэхгүй байгаа учраас цаашид энэ замаар яваад үр дүнд хүрэх эсэхэд итгэл алдрах болсон үед АХУСК (GIANTC)-ын 293 эрдэмтний хамтарсан нүсэр том судалгаа хариу өгөв (Allen H.L. нар 2010). Allen H.L. нар юуны өмнө ГХУ-ны судалгаа биеийн өндөр-уялдаат генийн биологийн үүрэг ажил, байгалийн жамыг илрүүлж танин мэдэхэд нэн чухал үнэт арга зүй мөн болохыг дахин нотлов. Тэд нийт 46 судалгааны 183’727 хүний дүнг нэгтгэсэн дам-анализ хийж биеийн өндөртэй P <5 x 10 -8 нарийвчлал бүхий уялдаатай 180 локус олж тогтоосон байна. Шинээр нээсэн эдгээр олон локус хүний төрөл бүрийн өвчин болон тоон хэмжээст зүстөрхийн удамзүйн судалгаанд чухал ач холбогдолтой олон үзэгдлийн нээн илрүүлжээ. Тухайлбал, эдгээр 180 локус санамсаргүй тохиолдлоор олон газар, олон хромосом дээр тарж байрласан биш ажээ. Харин ч эсрэгээрээ биологийн үүрэг жамын ондоошил бүхий бусад олон генийн дотор, эсвэл хүрээлэлд, эсвэл ойролцоо оршиж тэдгээр генийн үйл ажиллагаагаар баяждаг байна. Тухайлбал, биеийн өндөр-уялдаат 180 вариант тус бүрийн үүрэг ажлын хоёр төрөл болох ижил-бус ННП (ибННП, англ. nsSNPs-non-synonynymous SNPs) ба цис-илрэх тоон хэмжээст локус (цис-иТХЛ, англ. cis-eQTL-cis-expression quantitative trait loci) хоорондын давхцааг анх туршиж биеийн өндөр-уялдаат локусууд цис-иТХЛ-тай хавьгүй их буюу 2,4 дахин олон (47 вариант : P=4,7 x 10 -11), ибННП-тай 1,7 дахин олон бөгөөд нягт корреляцитай (r2>= 0,8 HapMap Consortium) давхцаж өөрийн генийнхээ үүрэг ажлыг баяжуулан уургийн амин хүчлийн бүтцийг өөрчлөх вариантыг давуу төлөөлж зэргэлдээх генийн илрэлийг нэмэгдүүлж байжээ. Тодорхой локус дээр цис-иТХЛ эсвэл ибННП уялдаат локустай давхцан оршиж баяжуулж байгаа нь нийтлэг үүрэг ажлын вариантууд хүчтэй-уялдаат локус дахь шалтгаан үрийн вариантын илрэлд оролцдогийг харуулж байна. Мөн эдгээр шинээр нээсэн локусууд ба хүний араг ясны өсөлтийн согогийн хамшинжээр илэрдэг генийн мутацууд хоорондоо холбоотой эсэхийг судалж 180 локусын 21 вариант энэ төрлийн мутац өгдөг генийн цомогтой холбоотой, үүнээс 13 ген уялдаат вариантад хамгийн ойр зайд оршиж байгааг тогтоожээ. Allen H.L. нар (2010) энэ судалгааныхаа явцад шинээр нээсэн өндөр-уялдаат локусуудын олон гишүүнт (хетероген) чанарыг судалж үзэхэд 180 локус дотроос багаар тооцоход 19 локус үл хамааралт олон вариант агуулж байжээ. Энэ нь биеийн өндрийн полиген шинжийн аллелиүд олон гишүүнт чанартайг гэрчилж одоогоор нээгдээд буй локусуудыг бүх талаас нь дахин нарийвчлан судлаж тухайн локус дахь нэмэлт вариантуудыг бүрэн илрүүлэх мөн уялдаат локусын нэмэлт фракцыг шинээр тогтоох шаардлагатайг харуулсан байна. Мөн 180 локус доторхи генүүдийн хооронд биологийн холбоос буй эсэхийг тогтоохын тулд хоёр арга хэрэглэсэн байна. Нэгдүгээрт, мэдээллийн далай дотроос үг шүүж тооцоолдог “Мэдэх Локус Хоорондын Генийн Харилцаа” (МЛХГХ, англ. GRAIL-Gene Relationship Among Implicated Loci) аргаар судалж үзэхэд 42 ген бусад локустай холбоотой байжээ.
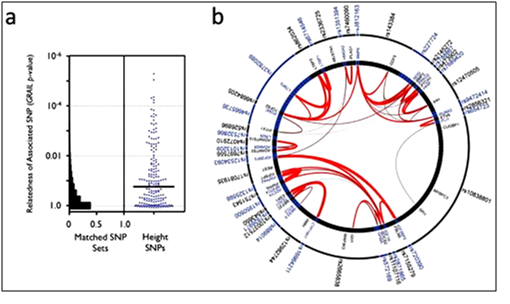
Зураг 8. Хоорондоо холбоотой ген агуулсан биеийн өндөрийн локусууд
(a) Өндөр-уялдаат 180 ННП. Босоо y-тэнхлэг дээр GRAIL аргаар тооцсон Р-утга (логарифмын шатлалаар). Диаграммын хар сүүдэр 1000-аар багцалсан 180 уялдаат ННП-ийн тооцсон GRAIL Р-утгын тархалт. Таруу хөх толбо өндрийн 180 ННП-ийн тус бүрийн GRAIL Р-утгын тархалт. Хөндлөн хар зураас GRAIL Р-утгын дундач (P=0.14). Генүүдийг холбосон толгой 10 үг: ‘өсөлт’, ‘киназ’, ‘фактор’, ‘хуулбарлал’, ‘дохиол’, ‘холбоос’, ‘ялгарал’, ‘хөгжил’, ‘инсулин’, ‘яс’. (b) ННП-ийн холбоосыг харуулсан график. GRAIL Р<0.01 утга бүхий 42 ННП-ийн тохирох генүүд. Бүдүүн улаан зураас хүчтэй холбоосыг заана.
Тэдгээрийн дотроос сэвлэг дохиолын ген, TGF-![]() , өсөлтийн дааврын генүүд хоорондоо хүчтэй холбоостой байжээ. Хоёрдугаарт, генийн бүрдэл баяжихын анализ (ГББА, англ. GSEA-gene set enrichment analysis) буюу уялдаат вариантын генийн бүрдэл баяжихын дам-анализ (УВГББДА, англ. MAGENDA-Meta-Analysis Gene-set Enrichment of variaNT Associations) аргаар судлахад биологийн 17 жам, молекулын 14 үүрэг ажил биеийн өндөр-уялдаат генийг баяжуулж буйг илрүүлжээ.
, өсөлтийн дааврын генүүд хоорондоо хүчтэй холбоостой байжээ. Хоёрдугаарт, генийн бүрдэл баяжихын анализ (ГББА, англ. GSEA-gene set enrichment analysis) буюу уялдаат вариантын генийн бүрдэл баяжихын дам-анализ (УВГББДА, англ. MAGENDA-Meta-Analysis Gene-set Enrichment of variaNT Associations) аргаар судлахад биологийн 17 жам, молекулын 14 үүрэг ажил биеийн өндөр-уялдаат генийг баяжуулж буйг илрүүлжээ.
Энэ судалгааны явцад одоогоор нээгдээд буй сул нөлөөт олон вариантын нөлөөний хэмжээ ба уялдааны хүчийг ашиглаад тооцоолоход биеийн өндрийн өвлөгдөх шинжийн ~20%-ийг нөхцөлдүүлдэг 697 локус байх ёстой ба судалгаанд 500’000 хүн хамруулбал эдгээр локусын 99,5%-ийг тодорхойлж чадах тооцоо гарчээ.
Биеийн өндөр зүстөрхийн удамзүйн үндсийг судалсан хэвлэлийн эх сурвалжийн дүгнэлт:
1. Хүний өндөр зүстөрх олон хүчин зүйлээс хамаарах боловч 80-90 хувийг хариуцан өвлүүлдэг удамшлын хүчин зүйлийг танин мэдэх зорилгоор эволюцийн дүнд тогтсон нийтлэг аллелийн давтамжтай ННП-ийн хаплотип дээр тулгуурласан зүүлт ННП генотайпингийн бүтээц ашиглаж хийсэн ГХУ-ны судалгааны дүнд биеийн өндөр-уялдаат удамшлын үндсийн ~10 орчим хувийг нөхцөлдүүлдэг 200 орчим сул нөлөөт локус, уялдаат ген, геномын төлөөллийг нээгээд байна. Цаашид нээгдээгүй байгаа биеийн өндрийн өвлөгдөх шинжийг илрүүлэхэд ховор аллелийн давтамжтай (ХАД, англ. MAF-minor allel frequency) ННП-ийн хаплотип дээр тулгуурласан зүүлт ННП генотайпингийн бүтээц ашиглан ГХУ-ны судалгаа өрнүүлэх шаардлагатай байна.
2. Биеийн өндөр-уялдаат сул нөлөөт олон локус олон гишүүнт хетероген шинжтэй байгаа нь одоогоор нээгдээд буй локусуудыг дахин судлаж тэдгээр локус дахь нэмэлт вариантуудыг бүрэн илрүүлэх, мэдэгдэж буй локусын нэмэлт фракцыг шинээр тогтоох шаардлагатай байна.
3. Биеийн өндрийн өвлөгдөх шинжийн удамшлын хүчин зүйлийн ихэнхи судалгаа европын уугуул цагаан арьст кавказойд элэнц угшилтай хүн амын бүлэг дээр хийгдсэн байна. Цаашид удам угсааны өөр угшил гаралтай хүн амын бүлэг дотор эволюцийн явцад бүрэлдэн тогтсон удамшлын нууцыг танин мэдэх, хүчтэй нөлөөт ген, геномын хэсгийг илрүүлэх шаардлагатай байна.
2. Silventoinen K, Sammalisto S, Perola M, Boomsma DI, Cornes BK, Davis C, Dunkel L, De Lange M, Harris JR, Hjelmborg JV, Luciano M, Martin NG, Mortensen J, Nisticò L, Pedersen NL, Skytthe A, Spector TD, Stazi MA, Willemsen G, Kaprio J (2003) Heritability of adult body height: a comparative study of twin cohorts in eight countries. Twin Res 6:399–408
3. Carmichael CM, McGue M (1995) A cross-sectional examination of height, weight, and body mass index in adult twins. J Gerontol A Biol Sci Med Sci 50:1995 B237–B244
4. Galton F. Regression towards mediocrity in hereditary stature. J R Antropol Inst 1885; 5:329-348
5. Pearson K, Lee A. On the laws of inheritance in man. I. Inheritance of physical characteristics. Biometrika 1903; 2:357-462
6. Fisher RA. The Correlation Between Relatives on the supposition of Mendelian Inheritance. Transactions of the Royal Society of Edinburgh 1918; 52:399-433.
7. Davey Smith G, Hart C, Upton M, Hole D, Gillis C, Watt G, Hawthorne V
Height and risk of death among men and women: aetiological implications of associations with cardiorespiratory disease and cancer mortality. 2000 Feb;54(2):97-103.
8. Gunnell D, Okasha M, Smith GD, Oliver SE, Sandhu J, Holly JM Height, leg length, and cancer risk: a systematic review. Department of Social Medicine, United Kingdom . 2001;23(2):313-42
9. Hirschhorn JN, Lindgren CM, Daly MJ, Kirby A, SchaVner SF, Burtt NP, Altshuler D, Parker A, Rioux JD, Platko J, Gaudet D, Hudson TJ, Groop LC, Lander ES (2001) Genomewide linkage analysis of stature in multiple populations reveals several regions with evidence of linkage to adult height. Am J Hum Genet 69:106–116
10. . Perola M, Ohman M, Hiekkalinna T, Leppävuori J, Pajukanta P, Wessman M, Koskenvuo M, Palotie A, Lange K, Kaprio J, Peltonen L (2001) Quantitative-trait-locus analysis of body-mass index and of stature, by combined analysis of genome scans of five Finnish study groups. Am J Hum Genet 69:117–123
11. Merikangas KR, Risch N. Genomic priorities and public health. Science 2003; 302:599-601
12. Risch N, Merikangas KR. The future of genetic Studies of complex human disease. Science 1996; 273:1516-1517
13. Risch NJ. Searching for genetic determinants in the new millennium. Nature 2000; 405:847-856
14. Lettre G, Butler JL, Ardlie KG, Hirschhorn JN. Common genetic variation in eight genes of the GH/IGF1 axis does not contribute to adult height variation Hum genet 2007 Sep;122(2):129-39. Epub 2007 Jun 2.
15. Frayling TM, Hattersley AT, McCarthy A, Holly J, Mitchell SM, Gloyn AL, Owen K, Davies D, Smith GD, Ben-Shlomo Y. A putative functional polymorphism in the IGF-I gene: association studies with type 2 diabetes, adult height, glucose tolerance, and fetal growth in U.K. populations. Diabetes 2002 Jul;51(7):2313-6.
16. Jorge AA, Marchisotti FG, Montenegro LR, Carvalho LR, Mendonca BB, Arnhold IJ. Growth hormone (GH) pharmacogenetics: influence of GH receptor exon 3 retention or deletion on first-year growth response and final height in patients with severe GH deficiency. J Clin Endocrinol Metab. 2006 Mar;91(3):1076-80. Epub 2005 Nov 15.
17. Hirschhorn J, Daly M. Genome-wide association studies for common diseases and complex traits. Nature Rev Genet 2005; 6:95-108
18. Wang WY, Barratt BJ, Clayton DG, Todd JA Genome-wide association studies: theoretical and practical concerns. Nat Rev Genet. 2005 Feb;6(2):109-18.
19. Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, Devon K, Dewar K, Doyle M, FitzHugh W, Funke R, Gage D, Harris K, Heaford A, Howland J,Kann L, Lehoczky J, LeVine R, McEwan P, McKernan K, Meldrim J, Mesirov JP, Miranda C, Morris W, Naylor J, Raymond C, Rosetti M, Santos R, Sheridan A,Sougnez C, Stange-Thomann N, Stojanovic N, Subramanian A, Wyman D, Rogers J, Sulston J, Ainscough R, Beck S, Bentley D, Burton J, Clee C, Carter N,Coulson A, Deadman R, Deloukas P, Dunham A, Dunham I, Durbin R, French L, Grafham D, Gregory S, Hubbard T, Humphray S, Hunt A, Jones M, Lloyd C,McMurray A, Matthews L, Mercer S, Milne S, Mullikin JC, Mungall A, Plumb R, Ross M, Shownkeen R, Sims S, Waterston RH, Wilson RK, Hillier LW, McPherson JD, Marra MA, Mardis ER, Fulton LA, Chinwalla AT, Pepin KH, Gish WR, Chissoe SL, Wendl MC, Delehaunty KD, Miner TL, Delehaunty A, Kramer JB, Cook LL,Fulton RS, Johnson DL, Minx PJ, Clifton SW, Hawkins T, Branscomb E, Predki P, Richardson P, Wenning S, Slezak T, Doggett N, Cheng JF, Olsen A, Lucas S,Elkin C, Uberbacher E, Frazier M, Gibbs RA, Muzny DM, Scherer SE, Bouck JB, Sodergren EJ, Worley KC, Rives CM, Gorrell JH, Metzker ML, Naylor SL,Kucherlapati RS, Nelson DL, Weinstock GM, Sakaki Y, Fujiyama A, Hattori M, Yada T, Toyoda A, Itoh T, Kawagoe C, Watanabe H, Totoki Y, Taylor T,Weissenbach J, Heilig R, Saurin W, Artiguenave F, Brottier P, Bruls T, Pelletier E, Robert C, Wincker P, Smith DR, Doucette-Stamm L, Rubenfield M, Weinstock K, Lee HM, Dubois J, Rosenthal A, Platzer M, Nyakatura G, Taudien S, Rump A, Yang H, Yu J, Wang J, Huang G, Gu J, Hood L, Rowen L, Madan A, Qin S, Davis RW, Federspiel NA, Abola AP, Proctor MJ, Myers RM, Schmutz J, Dickson M, Grimwood J, Cox DR, Olson MV, Kaul R, Raymond C, Shimizu N, Kawasaki K,Minoshima S, Evans GA, Athanasiou M, Schultz R, Roe BA, Chen F, Pan H, Ramser J, Lehrach H, Reinhardt R, McCombie WR, de la Bastide M, Dedhia N,Blöcker H, Hornischer K, Nordsiek G, Agarwala R, Aravind L, Bailey JA, Bateman A, Batzoglou S, Birney E, Bork P, Brown DG, Burge CB, Cerutti L, Chen HC,Church D, Clamp M, Copley RR, Doerks T, Eddy SR, Eichler EE, Furey TS, Galagan J, Gilbert JG, Harmon C, Hayashizaki Y, Haussler D, Hermjakob H,Hokamp K, Jang W, Johnson LS, Jones TA, Kasif S, Kaspryzk A, Kennedy S, Kent WJ, Kitts P, Koonin EV, Korf I, Kulp D, Lancet D, Lowe TM, McLysaght A,Mikkelsen T, Moran JV, Mulder N, Pollara VJ, Ponting CP, Schuler G, Schultz J, Slater G, Smit AF, Stupka E, Szustakowski J, Thierry-Mieg D, Thierry-Mieg J,Wagner L, Wallis J, Wheeler R, Williams A, Wolf YI, Wolfe KH, Yang SP, Yeh RF, Collins F, Guyer MS, Peterson J, Felsenfeld A, Wetterstrand KA, Patrinos A,Morgan MJ, de Jong P, Catanese JJ, Osoegawa K, Shizuya H, Choi S, Chen YJ; International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome. 2001 Feb 15;409(6822):860-921.
20. Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Huson DH,Wortman JR, Zhang Q, Kodira CD, Zheng XH, Chen L, Skupski M, Subramanian G, Thomas PD, Zhang J, Gabor Miklos GL, Nelson C, Broder S, Clark AG,Nadeau J, McKusick VA, Zinder N, Levine AJ, Roberts RJ, Simon M, Slayman C, Hunkapiller M, Bolanos R, Delcher A, Dew I, Fasulo D, Flanigan M, Florea L,Halpern A, Hannenhalli S, Kravitz S, Levy S, Mobarry C, Reinert K, Remington K, Abu-Threideh J, Beasley E, Biddick K, Bonazzi V, Brandon R, Cargill M,Chandramouliswaran I, Charlab R, Chaturvedi K, Deng Z, Di Francesco V, Dunn P, Eilbeck K, Evangelista C, Gabrielian AE, Gan W, Ge W, Gong F, Gu Z, Guan P, Heiman TJ, Higgins ME, Ji RR, Ke Z, Ketchum KA, Lai Z, Lei Y, Li Z, Li J, Liang Y, Lin X, Lu F, Merkulov GV, Milshina N, Moore HM, Naik AK, Narayan VA,Neelam B, Nusskern D, Rusch DB, Salzberg S, Shao W, Shue B, Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F, Zhong W, Zhu S, Zhao S, Gilbert D, Baumhueter S, Spier G, Carter C, Cravchik A, Woodage T, Ali F, An H, Awe A, Baldwin D, Baden H, Barnstead M, Barrow I, Beeson K, Busam D, Carver A, Center A, Cheng ML, Curry L, Danaher S, Davenport L, Desilets R, Dietz S,Dodson K, Doup L, Ferriera S, Garg N, Gluecksmann A, Hart B, Haynes J, Haynes C, Heiner C, Hladun S, Hostin D, Houck J, Howland T, Ibegwam C, Johnson J, Kalush F, Kline L, Koduru S, Love A, Mann F, May D, McCawley S, McIntosh T, McMullen I, Moy M, Moy L, Murphy B, Nelson K, Pfannkoch C, Pratts E, Puri V,Qureshi H, Reardon M, Rodriguez R, Rogers YH, Romblad D, Ruhfel B, Scott R, Sitter C, Smallwood M, Stewart E, Strong R, Suh E, Thomas R, Tint NN, Tse S,Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril JF, Guigó R, Campbell MJ, Sjolander KV, Karlak B, Kejariwal A, Mi H, Lazareva B, Hatton T, Narechania A, Diemer K, Muruganujan A, Guo N, Sato S, Bafna V, Istrail S, Lippert R, Schwartz R, Walenz B, Yooseph S, Allen D, Basu A, Baxendale J, Blick L, Caminha M, Carnes-Stine J, Caulk P, Chiang YH, Coyne M, Dahlke C, Mays A, Dombroski M, Donnelly M, Ely D,Esparham S, Fosler C, Gire H, Glanowski S, Glasser K, Glodek A, Gorokhov M, Graham K, Gropman B, Harris M, Heil J, Henderson S, Hoover J, Jennings D,Jordan C, Jordan J, Kasha J, Kagan L, Kraft C, Levitsky A, Lewis M, Liu X, Lopez J, Ma D, Majoros W, McDaniel J, Murphy S, Newman M, Nguyen T, Nguyen N,Nodell M, Pan S, Peck J, Peterson M, Rowe W, Sanders R, Scott J, Simpson M, Smith T, Sprague A, Stockwell T, Turner R, Venter E, Wang M, Wen M, Wu D, Wu M, Xia A, Zandieh A, Zhu X The sequence of the human genome. Science. 2001 Feb 16;291(5507):1304-51.
21. International HapMap Consortium. A haplotype map of the human genome. Nature 2005;437:1299-1320.
22. International HapMap Consortium, Frazer KA, Ballinger DG, Cox DR, Hinds DA, Stuve LL, Gibbs RA, Belmont JW, Boudreau A, Hardenbol P, Leal SM,Pasternak S, Wheeler DA, Willis TD, Yu F, Yang H, Zeng C, Gao Y, Hu H, Hu W, Li C, Lin W, Liu S, Pan H, Tang X, Wang J, Wang W, Yu J, Zhang B, Zhang Q,Zhao H, Zhao H, Zhou J, Gabriel SB, Barry R, Blumenstiel B, Camargo A, Defelice M, Faggart M, Goyette M, Gupta S, Moore J, Nguyen H, Onofrio RC, Parkin M,Roy J, Stahl E, Winchester E, Ziaugra L, Altshuler D, Shen Y, Yao Z, Huang W, Chu X, He Y, Jin L, Liu Y, Shen Y, Sun W, Wang H, Wang Y, Wang Y, Xiong X, Xu L, Waye MM, Tsui SK, Xue H, Wong JT, Galver LM, Fan JB, Gunderson K, Murray SS, Oliphant AR, Chee MS, Montpetit A, Chagnon F, Ferretti V, Leboeuf M,Olivier JF, Phillips MS, Roumy S, Sallée C, Verner A, Hudson TJ, Kwok PY, Cai D, Koboldt DC, Miller RD, Pawlikowska L, Taillon-Miller P, Xiao M, Tsui LC, Mak W, Song YQ, Tam PK, Nakamura Y, Kawaguchi T, Kitamoto T, Morizono T, Nagashima A, Ohnishi Y, Sekine A, Tanaka T, Tsunoda T, Deloukas P, Bird CP,Delgado M, Dermitzakis ET, Gwilliam R, Hunt S, Morrison J, Powell D, Stranger BE, Whittaker P, Bentley DR, Daly MJ, de Bakker PI, Barrett J, Chretien YR,Maller J, McCarroll S, Patterson N, Pe'er I, Price A, Purcell S, Richter DJ, Sabeti P, Saxena R, Schaffner SF, Sham PC, Varilly P, Altshuler D, Stein LD, Krishnan L, Smith AV, Tello-Ruiz MK, Thorisson GA, Chakravarti A, Chen PE, Cutler DJ, Kashuk CS, Lin S, Abecasis GR, Guan W, Li Y, Munro HM, Qin ZS, Thomas DJ,McVean G, Auton A, Bottolo L, Cardin N, Eyheramendy S, Freeman C, Marchini J, Myers S, Spencer C, Stephens M, Donnelly P, Cardon LR, Clarke G, Evans DM, Morris AP, Weir BS, Tsunoda T, Mullikin JC, Sherry ST, Feolo M, Skol A, Zhang H, Zeng C, Zhao H, Matsuda I, Fukushima Y, Macer DR, Suda E, Rotimi CN,Adebamowo CA, Ajayi I, Aniagwu T, Marshall PA, Nkwodimmah C, Royal CD, Leppert MF, Dixon M, Peiffer A, Qiu R, Kent A, Kato K, Niikawa N, Adewole IF,Knoppers BM, Foster MW, Clayton EW, Watkin J, Gibbs RA, Belmont JW, Muzny D, Nazareth L, Sodergren E, Weinstock GM, Wheeler DA, Yakub I, Gabriel SB,Onofrio RC, Richter DJ, Ziaugra L, Birren BW, Daly MJ, Altshuler D, Wilson RK, Fulton LL, Rogers J, Burton J, Carter NP, Clee CM, Griffiths M, Jones MC, McLay K, Plumb RW, Ross MT, Sims SK, Willey DL, Chen Z, Han H, Kang L, Godbout M, Wallenburg JC, L'Archevêque P, Bellemare G, Saeki K, Wang H, An D, Fu H,Li Q, Wang Z, Wang R, Holden AL, Brooks LD, McEwen JE, Guyer MS, Wang VO, Peterson JL, Shi M, Spiegel J, Sung LM, Zacharia LF, Collins FS, Kennedy K,Jamieson R, Stewart J. A second generation human haplotype map of over 3.1 million SNPs. Nature. 2007 Oct 18;449(7164):851-61.
23. Korn JM, Kuruvilla FG, McCarroll SA, Wysoker A, Nemesh J, Cawley S, Hubbell E, Veitch J, Collins PJ, Darvishi K, Lee C, Nizzari MM, Gabriel SB, Purcell S, Daly MJ, Altshuler D. Integrated genotype calling and association analysis of SNPs, common copy number polymorphisms and rare CNVs. Nat Genet. 2008 Oct;40(10):1253-60.
24. McCarroll SA, Kuruvilla FG, Korn JM, Cawley S, Nemesh J, Wysoker A, Shapero MH, de Bakker PI, Maller JB, Kirby A, Elliott AL, Parkin M, Hubbell E, Webster T,Mei R, Veitch J, Collins PJ, Handsaker R, Lincoln S, Nizzari M, Blume J, Jones KW, Rava R, Daly MJ, Gabriel SB, Altshuler D. Integrated detection and population-genetic analysis of SNPs and copy number variation. Nat Genet. 2008 Oct;40(10):1166-74.
25. Pe'er I, de Bakker PI, Maller J, Yelensky R, Altshuler D, Daly MJ. Evaluating and improving power in whole-genome association studies using fixed marker sets. Nat Genet. 2006 Jun;38(6):663-7.
26. Lohmueller KE, Pearce CL, Pike M, Lander ES, Hirschhorn JN. Meta-analysis of genetic association studies supports a contribution of common variants to susceptibility to common disease. Nat Genet. 2003 Feb;33(2):177-82.
27. McCarthy MI, Abecasis GR, Cardon LR, Goldstein DB, Little J, Ioannidis JP, Hirschhorn JN. Genome-wide association studies for complex traits: consensus, uncertainty and challenges. Nat Rev Genet. 2008 May;9(5):356-69
28. The Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common disease and 3,000 shared controls. Nature 2007; 447:661-678
29. Clayton DG, Walker NM, Smyth DJ, Pask R, Cooper JD, Maier LM, Smink LJ, Lam AC, Ovington NR, Stevens HE, Nutland S, Howson JM, Faham M, Moorhead M, Jones HB, Falkowski M, Hardenbol P, Willis TD, Todd JA. Population structure, differential bias and genomic control in a large-scale, case-control association study. Nat Genet. 2005 Nov;37(11):1243-6
30. Campbell CD, Ogburn EL, Lunetta KL, Lyon HN, Freedman ML, Groop LC, Altshuler D, Ardlie KG, Hirschhorn JN. Demonstrating stratification in a European American population. Nat Genet. 2005 Aug;37(8):868-72.
31. Price AL, Patterson NJ, Plenge RM, Weinblatt ME, Shadick NA, Reich D. Principal components analysis corrects for stratification in genome-wide association studies. Nat Genet. 2006 Aug;38(8):904-909
32. Price AL, Butler J, Patterson N, Capelli C, Pascali VL, Scarnicci F, Ruiz-Linares A, Groop L, Saetta AA, Korkolopoulou P, Seligsohn U, Waliszewska A, Schirmer C, Ardlie K, Ramos A, Nemesh J, Arbeitman L, Goldstein DB, Reich D, Hirschhorn JN. Discerning the ancestry of European Americans in genetic association studies. PLoS Genet. 2008 Jan;4(1):e236.
33. Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, Maller J, Sklar P, de Bakker PI, Daly MJ, Sham PC. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 2007 Sep;81(3):559-75
34. Abecasis GR, Cherny SS, Cookson WO, Cardon LR. Merlin--rapid analysis of dense genetic maps using sparse gene flow trees. Nat Genet. 2002 Jan;30(1):97-101.
35. Saxena R, Voight BF,Lyssenko V, Burtt NP, de Bakker PI, Chen H, Roix JJ, Kathiresan S, Hirschhorn JN, Daly MJ, Hughes TE, Groop L, Altshuler D, Almgren P, Florez JC, Meyer J,Ardlie K, Bengtsson Boström K, Isomaa B, Lettre G, Lindblad U, Lyon HN, Melander O, Newton-Cheh C, Nilsson P, Orho-Melander M, Råstam L, Speliotes EK,Taskinen MR, Tuomi T, Guiducci C, Berglund A, Carlson J, Gianniny L, Hackett R, Hall L, Holmkvist J, Laurila E, Sjögren M, Sterner M, Surti A, Svensson M,Svensson M, Tewhey R, Blumenstiel B, Parkin M, Defelice M, Barry R, Brodeur W, Camarata J, Chia N, Fava M, Gibbons J, Handsaker B, Healy C, Nguyen K,Gates C, Sougnez C, Gage D, Nizzari M, Gabriel SB, Chirn GW, Ma Q, Parikh H, Richardson D, Ricke D, Purcell S. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels.Science 2007; 316:1331-1336.
36. Zeggini E, Weedon MN, Lindgren CM, Frayling TM, Elliott KS, Lango H, Timpson NJ, Perry JR, Rayner NW, Freathy RM, Barrett JC, Shields B, Morris AP, Ellard S, Groves CJ, Harries LW, Marchini JL, Owen KR, Knight B, Cardon LR, Walker M, Hitman GA, Morris AD, Doney AS; Wellcome Trust Case Control Consortium (WTCCC), McCarthy MI, Hattersley AT. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science. 2007 Aug 24;317(5841):1035-6.
37. Weedon MN, Lettre G, Freathy RM, Lindgren CM, Voight BF, Perry JR, Elliott KS, Hackett R, Guiducci C, Shields B, Zeggini E, Lango H, Lyssenko V, Timpson NJ, Burtt NP, Rayner NW, Saxena R, Ardlie K, Tobias JH, Ness AR, Ring SM, Palmer CN, Morris AD, Peltonen L, Salomaa V; Diabetes Genetics Initiative; Wellcome Trust Case Control Consortium, Davey Smith G, Groop LC, Hattersley AT, McCarthy MI, Hirschhorn JN, Frayling TM. A common variant of HMGA2 is associated with adult and childhood height in the general population.Nat Genet 2007 Oct;39(10):1245-50. Epub 2007 Sep 2.
38. Mayr C, Hemann MT, Bartel Disrupting the pairing between let-7 and Hmga2 enhances oncogenic transformation.DP Science 2007 Mar 16;315(5818):1576-9. Epub 2007 Feb 22.
39. Zhou X, Benson KF, Ashar HR, Chada K. Mutation responsible for the mouse pygmy phenotype in the developmentally regulated factor HMGI-C. Nature 1995 Aug 31;376(6543):771-4
40. Ligon AH, Moore SD, Parisi MA, Mealiffe ME, Harris DJ, Ferguson HL, Quade BJ, Morton CC. Constitutional rearrangement of the architectural factor HMGA2: a novel human phenotype including overgrowth and lipomas. Hum Genet 2005 Feb;76(2):340-8. Epub 2004 Dec 10.
41. Menten B, Buysse K, Zahir F, Hellemans J, Hamilton SJ, Costa T, Fagerstrom C, Anadiotis G, Kingsbury D, McGillivray BC, Marra MA, Friedman JM, Speleman F, Mortier G Osteopoikilosis, short stature and mental retardation as key features of a new microdeletion syndrome on 12q14. Med Jenet 2007 Apr;44(4):264-8. Epub 2007 Jan 12.
42. Sanna S, Jackson AU, Nagaraja R, Willer CJ, Chen WM, Bonnycastle LL, Shen H, Timpson N, Lettre G, Usala G, Chines PS, Stringham HM, Scott LJ, Dei M, Lai S, Albai G, Crisponi L, Naitza S, Doheny KF, Pugh EW, Ben-Shlomo Y, Ebrahim S, Lawlor DA, Bergman RN, Watanabe RM, Uda M, Tuomilehto J, Coresh J, Hirschhorn JN, Shuldiner AR, Schlessinger D, Collins FS, Davey Smith G, Boerwinkle E, Cao A, Boehnke M, Abecasis GR, Mohlke KL (2008) Common variants in the GDF5-UQCC region are associated with variation in human height. Nat Genet 40:198–203
43. Thomas JT, Lin K, Nandedkar M, Camargo M, Cervenka J, Luyten FP A human chondrodysplasia due to a mutation in a TGF-beta superfamily member.Nat Genet 1996 Mar;12(3):315-317.
44. Polinkovsky A, Robin NH, Thomas JT, Irons M, Lynn A, Goodman FR, Reardon W, Kant SG, Brunner HG, van der Burgt I, Chitayat D, McGaughran J, Donnai D,Luyten FP, Warman ML. Mutations in CDMP1 cause autosomal dominant brachydactyly type C.Nat Genet. 1997 Sep;17(1):18-9.
45. Miyamoto Y, Mabuchi A, Shi D, Kubo T, Takatori Y, Saito S, Fujioka M, Sudo A, Uchida A, Yamamoto S, Ozaki K, Takigawa M, Tanaka T, Nakamura Y, Jiang Q,Ikegawa S. A functional polymorphism in the 5' UTR of GDF5 is associated with susceptibility to osteoarthritis. Nat Genet. 2007 Apr;39(4):529-33.
46. Gudbjartsson DF, Walters GB, Thorleifsson G, Stefansson H, Halldorsson BV, Zusmanovich P, Sulem P, Thorlacius S, Gylfason A, Steinberg S, Helgadottir A, Ingason A, Steinthorsdottir V, Olafsdottir EJ, Olafsdottir GH, Jonsson T, Borch-Johnsen K, Hansen T,Andersen G, Jorgensen T, Pedersen O, Aben KK, Witjes JA,Swinkels DW, den Heijer M, Franke B, Verbeek AL, Becker DM,Yanek LR, Becker LC, Tryggvadottir L, Rafnar T, Gulcher J, KiemeneyLA, Kong A, Thorsteinsdottir U, Stefansson K (2008) Many sequence variants aVecting diversity of adult human height.Nat Genet 40:609–615
47. Lettre G, Jackson AU, Gieger C, Schumacher FR, Berndt SI, Sanna S, Eyheramendy S, Voight BF, Butler JL, Guiducci C, Illig T, Hackett R, Heid IM, Jacobs KB, Lyssenko V, Uda M; The Diabetes Genetics Initiative; FUSION; KORA; The Prostate, Lung Colorectal and Ovarian Cancer Screening Trial; The Nurses’ Health Study; SardiNIA, Boehnke M, Chanock SJ, Groop LC, Hu FB, Isomaa B, Kraft P, Peltonen L, Salomaa V, Schlessinger D, Hunter DJ, Hayes RB, Abecasis GR, Wichmann HE, Mohlke KL, Hirschhorn JN (2008) Identiffcation of ten loci associated with height highlights new biological pathways in human growth. Nat Genet. 40:584–591
48. Weedon MN, Lango H, Lindgren CM, Wallace C, Evans DM, Mangino M, Freathy RM, Perry JR, Stevens S, Hall AS, Samani NJ, Shields B, Prokopenko I, Farrall M, Dominiczak A; Diabetes Genetics Initiative; Wellcome Trust Case Control Consortium, Johnson T, Bergmann S, Beckmann JS, Vollenweider P, Waterworth DM, Mooser V, Palmer CN, Morris AD, Ouwehand WH; Cambridge GEM Consortium, Zhao JH, Li S, Loos RJ, Barroso I, Deloukas P, Sandhu MS, Wheeler E, Soranzo N, Inouye M, Wareham NJ, CaulWeld M, Munroe PB, Hattersley AT, McCarthy MI, Frayling TM (2008) Genome-wide association analysis identiWes 20 loci that inXuence adult height. Nat Genet. 40:575–583
49. Johansson A, Marroni F, Hayward C, Franklin CS, Kirichenko AV, Jonasson I, Hicks AA, Vitart V, Isaacs A, Axenovich T, Campbell S, Dunlop MG, Floyd J, Hastie N, Hofman A, Knott S, Kolcic I, Pichler I, Polasek O, Rivadeneira F, Tenesa A, Uitterlinden AG, Wild SH, Zorkoltseva IV, Meitinger T, Wilson JF, Rudan I,Campbell H, Pattaro C, Pramstaller P, Oostra BA, Wright AF, van Duijn CM, Aulchenko YS, Gyllensten U; EUROSPAN Consortium. Common variants in the JAZF1 gene associated with height identified by linkage and genome-wide association analysis. Hum Mol Genet. 2009 Jan 15;18(2):373-80.
50. Soranzo N, Rivadeneira F Chinappen-Horsley U, Malkina I, Richards JB, Hammond N, Stolk L, Nica A, Inouye M, Hofman A, Stephens J, Wheeler E, Arp P, Gwilliam R, Jhamai PM, Potter S, Chaney A, Ghori MJ, Ravindrarajah R, Ermakov S, Estrada K, Pols HA, Williams FM, McArdle WL, van Meurs JB, Loos RJ, Dermitzakis ET, Ahmadi KR, Hart DJ, Ouwehand WH, Wareham NJ, Barroso I, Sandhu MS, Strachan DP, Livshits G, Spector TD, Uitterlinden AG, Deloukas P.Meta-analysis of genome-wide scans for human adult stature identifies novel Loci and associations with measures of skeletal frame size. PLoS Genet. 2009 Apr;5(4):e1000445.
51. Maher B. Personal genomes: The case of the missing heritability. Nature 2008; 456:18-21
52. Zeggini E, Scott LJ, Saxena R, Voight BF, Marchini JL, Hu T, de Bakker PI, Abecasis GR, Almgren P, Andersen G, Ardlie K, Boström KB, Bergman RN,Bonnycastle LL, Borch-Johnsen K, Burtt NP, Chen H, Chines PS, Daly MJ, Deodhar P, Ding CJ, Doney AS, Duren WL, Elliott KS, Erdos MR, Frayling TM, Freathy RM, Gianniny L, Grallert H, Grarup N, Groves CJ, Guiducci C, Hansen T, Herder C, Hitman GA, Hughes TE, Isomaa B, Jackson AU, Jørgensen T, Kong A,Kubalanza K, Kuruvilla FG, Kuusisto J, Langenberg C, Lango H, Lauritzen T, Li Y, Lindgren CM, Lyssenko V, Marvelle AF, Meisinger C, Midthjell K, Mohlke KL,Morken MA, Morris AD, Narisu N, Nilsson P, Owen KR, Palmer CN, Payne F, Perry JR, Pettersen E, Platou C, Prokopenko I, Qi L, Qin L, Rayner NW, Rees M,Roix JJ, Sandbaek A, Shields B, Sjögren M, Steinthorsdottir V, Stringham HM, Swift AJ, Thorleifsson G, Thorsteinsdottir U, Timpson NJ, Tuomi T, Tuomilehto J,Walker M, Watanabe RM, Weedon MN, Willer CJ; Wellcome Trust Case Control Consortium, Illig T, Hveem K, Hu FB, Laakso M, Stefansson K, Pedersen O,Wareham NJ, Barroso I, Hattersley AT, Collins FS, Groop L, McCarthy MI, Boehnke M, Altshuler D. Meta-analysis of genome-wide association data and large-scale replication identifies additional susceptibility loci for type 2 diabetes.Nat Genet. 2008 May;40(5):638-45. 53. Thomas G, Jacobs KB, Yeager M, Kraft P, Wacholder S, Orr N, Yu K, Chatterjee N, Welch R, Hutchinson A, Crenshaw A, Cancel-Tassin G, Staats BJ, Wang Z,Gonzalez-Bosquet J, Fang J, Deng X, Berndt SI, Calle EE, Feigelson HS, Thun MJ, Rodriguez C, Albanes D, Virtamo J, Weinstein S, Schumacher FR,Giovannucci E, Willett WC, Cussenot O, Valeri A, Andriole GL, Crawford ED, Tucker M, Gerhard DS, Fraumeni JF Jr, Hoover R, Hayes RB, Hunter DJ, Chanock SJ. Multiple loci identified in a genome-wide association study of prostate cancer. Nat Genet. 2008 Mar;40(3):310-5. 54. Smits P, Bolton AD, Funari V, Hong M, Boyden ED, Lu L, Manning DK, Dwyer ND, Moran JL, Prysak M, Merriman B, Nelson SF, Bonafé L, Superti-Furga A,Ikegawa S, Krakow D, Cohn DH, Kirchhausen T, Warman ML, Beier DR. Lethal skeletal dysplasia in mice and humans lacking the golgin GMAP-210. N Engl J Med. 2010 Jan 21;362(3):206-16.
55. Binder G, Seidel AK, Martin DD, Schweizer R, Schwarze CP, Wollmann HA, Eggermann T, Ranke MB. The endocrine phenotype in silver-russell syndrome is defined by the underlying epigenetic alteration. J Clin Endocrinol Metab. 2008 Apr;93(4):1402-7.
56. lettre G. Genetic regulation of adult stature. 2009, 21:515-522
57. Filion GJ, Zhenilo S, Salozhin S, Yamada D, Prokhortchouk E, Defossez PA A family of human zinc finger proteins that bind methylated DNA and repress transcription. Mol Cell Biol. 2006 Jan;26(1):169-81.
58.. Lango Allen H, Estrada K, Lettre G, Berndt SI, Weedon MN, Rivadeneira F, Willer CJ, Jackson AU, Vedantam S, Raychaudhuri S, Ferreira T, Wood AR, Weyant RJ, Segrè AV, Speliotes EK, Wheeler E, Soranzo N, Park JH, Yang J, Gudbjartsson D, Heard-Costa NL, Randall JC, Qi L, Vernon Smith A, Mägi R, Pastinen T,Liang L, Heid IM, Luan J, Thorleifsson G, Winkler TW, Goddard ME, Sin Lo K, Palmer C, Workalemahu T, Aulchenko YS, Johansson A, Zillikens MC, Feitosa MF, Esko T, Johnson T, Ketkar S, Kraft P, Mangino M, Prokopenko I, Absher D, Albrecht E, Ernst F, Glazer NL, Hayward C, Hottenga JJ, Jacobs KB, Knowles JW,Kutalik Z, Monda KL, Polasek O, Preuss M, Rayner NW, Robertson NR, Steinthorsdottir V, Tyrer JP, Voight BF, Wiklund F, Xu J, Zhao JH, Nyholt DR, Pellikka N,Perola M, Perry JR, Surakka I, Tammesoo ML, Altmaier EL, Amin N, Aspelund T, Bhangale T, Boucher G, Chasman DI, Chen C, Coin L, Cooper MN, Dixon AL,Gibson Q, Grundberg E, Hao K, Juhani Junttila M, Kaplan LM, Kettunen J, König IR, Kwan T, Lawrence RW, Levinson DF, Lorentzon M, McKnight B, Morris AP,Müller M, Suh Ngwa J, Purcell S, Rafelt S, Salem RM, Salvi E, Sanna S, Shi J, Sovio U, Thompson JR, Turchin MC, Vandenput L, Verlaan DJ, Vitart V, White CC,Ziegler A, Almgren P, Balmforth AJ, Campbell H, Citterio L, De Grandi A, Dominiczak A, Duan J, Elliott P, Elosua R, Eriksson JG, Freimer NB, Geus EJ, Glorioso N, Haiqing S, Hartikainen AL, Havulinna AS, Hicks AA, Hui J, Igl W, Illig T, Jula A, Kajantie E, Kilpeläinen TO, Koiranen M, Kolcic I, Koskinen S, Kovacs P,Laitinen J, Liu J, Lokki ML, Marusic A, Maschio A, Meitinger T, Mulas A, Paré G, Parker AN, Peden JF, Petersmann A, Pichler I, Pietiläinen KH, Pouta A,Ridderstråle M, Rotter JI, Sambrook JG, Sanders AR, Schmidt CO, Sinisalo J, Smit JH, Stringham HM, Bragi Walters G, Widen E, Wild SH, Willemsen G,Zagato L, Zgaga L, Zitting P, Alavere H, Farrall M, McArdle WL, Nelis M, Peters MJ, Ripatti S, van Meurs JB, Aben KK, Ardlie KG, Beckmann JS, Beilby JP,Bergman RN, Bergmann S, Collins FS, Cusi D, den Heijer M, Eiriksdottir G, Gejman PV, Hall AS, Hamsten A, Huikuri HV, Iribarren C, Kähönen M, Kaprio J,Kathiresan S, Kiemeney L, Kocher T, Launer LJ, Lehtimäki T, Melander O, Mosley TH Jr, Musk AW, Nieminen MS, O'Donnell CJ, Ohlsson C, Oostra B, Palmer LJ, Raitakari O, Ridker PM, Rioux JD, Rissanen A, Rivolta C, Schunkert H, Shuldiner AR, Siscovick DS, Stumvoll M, Tönjes A, Tuomilehto J, van Ommen GJ,Viikari J, Heath AC, Martin NG, Montgomery GW, Province MA, Kayser M, Arnold AM, Atwood LD, Boerwinkle E, Chanock SJ, Deloukas P, Gieger C, Grönberg H,Hall P, Hattersley AT, Hengstenberg C, Hoffman W, Lathrop GM, Salomaa V, Schreiber S, Uda M, Waterworth D, Wright AF, Assimes TL, Barroso I, Hofman A,Mohlke KL, Boomsma DI, Caulfield MJ, Cupples LA, Erdmann J, Fox CS, Gudnason V, Gyllensten U, Harris TB, Hayes RB, Jarvelin MR, Mooser V, Munroe PB,Ouwehand WH, Penninx BW, Pramstaller PP, Quertermous T, Rudan I, Samani NJ, Spector TD, Völzke H, Watkins H, Wilson JF, Groop LC, Haritunians T, Hu FB, Kaplan RC, Metspalu A, North KE, Schlessinger D, Wareham NJ, Hunter DJ, O'Connell JR, Strachan DP, Wichmann HE, Borecki IB, van Duijn CM, Schadt EE, Thorsteinsdottir U, Peltonen L, Uitterlinden AG, Visscher PM, Chatterjee N, Loos RJ, Boehnke M, McCarthy MI, Ingelsson E, Lindgren CM, Abecasis GR,Stefansson K, Frayling TM, Hirschhorn JN. Hundreds of variants clustered in genomic loci and biological pathways affect human height. Nature. 2010 Oct 14;467(7317):832-838




